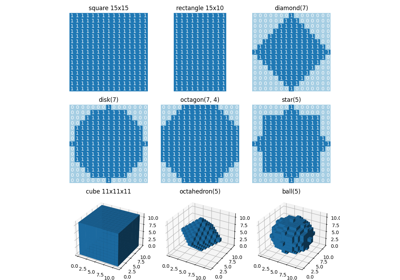
skimage.morphology#
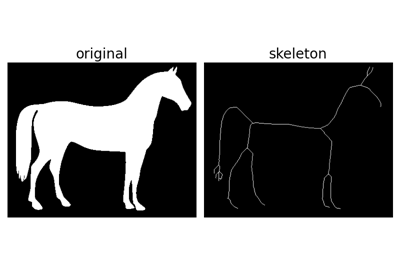
形態學演算法,例如:閉合、開啟、骨骼化。
執行影像的面積閉合。 |
|
執行影像的面積開啟。 |
|
產生球狀的足跡。 |
|
回傳影像的快速二元形態學閉合。 |
|
回傳影像的快速二元形態學膨脹。 |
|
回傳影像的快速二元形態學侵蝕。 |
|
回傳影像的快速二元形態學開啟。 |
|
回傳影像的黑色禮帽。 |
|
回傳影像的灰階形態學閉合。 |
|
計算二元影像的凸包影像。 |
|
計算二元影像中個別物件的凸包影像。 |
|
執行影像的直徑閉合。 |
|
執行影像的直徑開啟。 |
|
產生平坦的菱形足跡。 |
|
回傳影像的灰階形態學膨脹。 |
|
產生平坦的圓盤狀足跡。 |
|
產生平坦的橢圓形足跡。 |
|
回傳影像的灰階形態學侵蝕。 |
|
對應到洪水填充的遮罩。 |
|
在影像上執行洪水填充。 |
|
將足跡序列轉換為等效的 ndarray。 |
|
產生矩形或超矩形足跡。 |
|
決定影像中所有高度 >= h 的最大值。 |
|
決定影像中所有深度 >= h 的最小值。 |
|
回傳影像的二元形態學閉合。 |
|
回傳影像的二元形態學膨脹。 |
|
回傳影像的二元形態學侵蝕。 |
|
回傳影像的二元形態學開啟。 |
|
標記整數陣列的連通區域。 |
|
尋找 n 維陣列的局部最大值。 |
|
尋找 n 維陣列的局部最小值。 |
|
從影像建立最大樹。 |
|
決定影像的所有局部最大值。 |
|
計算二元影像的中軸轉換。 |
|
鏡像足跡中的每個維度。 |
|
產生八邊形足跡。 |
|
產生八面體足跡。 |
|
回傳影像的灰階形態學開啟。 |
|
沿著每個維度將足跡填充為奇數大小。 |
|
執行影像的形態學重建。 |
|
按照指定的順序移除物件,直到剩餘物件保持最小距離。 |
|
移除小於指定大小的連續孔洞。 |
|
移除小於指定大小的物件。 |
|
透過細化計算輸入影像的骨骼。 |
|
產生星形足跡。 |
|
執行二元影像的形態學細化。 |
|
回傳影像的白色禮帽。 |
- skimage.morphology.area_closing(image, area_threshold=64, connectivity=1, parent=None, tree_traverser=None)[原始碼]#
執行影像的面積閉合。
面積閉合會移除影像中所有表面小於 area_threshold 的暗色結構。輸出影像對於每個像素都大於或等於輸入影像,並且所有局部最小值都至少具有 area_threshold 像素的表面。
面積閉合類似於形態學閉合,但它們不使用固定的足跡,而是使用可變形的足跡,其表面 = area_threshold。
在二元情況下,面積閉合等同於 remove_small_holes;因此此運算子會擴展到灰階影像。
從技術上講,此運算子基於影像的最大樹表示。
- 參數:
- imagendarray
要計算面積閉合的輸入影像。此影像可以是任何類型。
- area_threshold無號整數
大小參數(像素數)。預設值任意選擇為 64。
- connectivity無號整數,選用
鄰域連通性。整數表示到達鄰居的最大正交步數。在 2D 中,4 鄰域為 1,而 8 鄰域為 2。預設值為 1。
- parentndarray, int64,選用
表示反轉影像的最大樹的父影像。每個像素的值是其在展開陣列中的父索引。請參閱「注意」以取得更多詳細資訊。
- tree_traverser1D 陣列,int64,選用
已排序的像素索引(參考展開的陣列)。像素的排序方式為每個像素都位於其父項之前(根除外,根沒有父項)。
- 回傳:
- outputndarray
輸出影像的形狀和類型與輸入影像相同。
另請參閱
注意
如果將最大樹表示法(父項和 tree_traverser)提供給函式,則必須從此函式的反轉影像計算它們,即:>>> P, S = max_tree(invert(f)) >>> closed = diameter_closing(f, 3, parent=P, tree_traverser=S)
參考文獻
[1]Vincent L., Proc. “灰階面積開啟和閉合,其有效實作和應用”,EURASIP 數學形態學及其在訊號處理的應用研討會,西班牙巴塞隆納,第 22-27 頁,1993 年 5 月。
[2]Soille, P., “形態學影像分析:原理和應用”(第 6 章),第 2 版 (2003),ISBN 3540429883。DOI:10.1007/978-3-662-05088-0
[3]Salembier, P., Oliveras, A., & Garrido, L. (1998)。用於影像和序列處理的非擴張連通運算子。IEEE 影像處理交易,7(4),555-570。DOI:10.1109/83.663500
[4]Najman, L., & Couprie, M. (2006)。以擬線性時間建立元件樹。IEEE 影像處理交易,15(11),3531-3539。DOI:10.1109/TIP.2006.877518
[5]Carlinet, E., & Geraud, T. (2014)。元件樹計算演算法的比較評論。IEEE 影像處理交易,23(9),3885-3895。DOI:10.1109/TIP.2014.2336551
範例
我們建立一個影像(在中心具有最小值和 4 個額外局部最小值的二次函數)。
>>> w = 12 >>> x, y = np.mgrid[0:w,0:w] >>> f = 180 + 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:3,1:5] = 160; f[2:4,9:11] = 140; f[9:11,2:4] = 120 >>> f[9:10,9:11] = 100; f[10,10] = 100 >>> f = f.astype(int)
我們可以計算面積閉合
>>> closed = area_closing(f, 8, connectivity=1)
移除所有小的最小值,剩下的最小值至少具有大小為 8。
- skimage.morphology.area_opening(image, area_threshold=64, connectivity=1, parent=None, tree_traverser=None)[原始碼]#
執行影像的面積開啟。
面積開啟會移除影像中所有表面小於 area_threshold 的亮色結構。因此,輸出影像是小於輸入且所有局部最大值至少具有 area_threshold 像素表面的最大影像。
面積開啟類似於形態學開啟,但它們不使用固定的足跡,而是使用可變形的足跡,其表面 = area_threshold。因此,area_opening 與 area_threshold=1 是恆等式。
在二元情況下,面積開啟等同於 remove_small_objects;因此此運算子會擴展到灰階影像。
從技術上講,此運算子基於影像的最大樹表示。
- 參數:
- imagendarray
要計算面積開啟的輸入影像。此影像可以是任何類型。
- area_threshold無號整數
大小參數(像素數)。預設值任意選擇為 64。
- connectivity無號整數,選用
鄰域連通性。整數表示到達鄰居的最大正交步數。在 2D 中,4 鄰域為 1,而 8 鄰域為 2。預設值為 1。
- parentndarray, int64,選用
父圖像,表示該影像的最大樹。每個像素的值是其在展開陣列中父節點的索引。
- tree_traverser1D 陣列,int64,選用
已排序的像素索引(參考展開的陣列)。像素的排序方式為每個像素都位於其父項之前(根除外,根沒有父項)。
- 回傳:
- outputndarray
輸出影像,與輸入影像具有相同的形狀和類型。
另請參閱
參考文獻
[1]Vincent L., Proc. “灰階面積開啟和閉合,其有效實作和應用”,EURASIP 數學形態學及其在訊號處理的應用研討會,西班牙巴塞隆納,第 22-27 頁,1993 年 5 月。
[2]Soille, P., “形態學影像分析:原理和應用”(第 6 章),第 2 版 (2003),ISBN 3540429883。DOI:10.1007/978-3-662-05088-0
[3]Salembier, P., Oliveras, A., & Garrido, L. (1998)。用於影像和序列處理的非擴張連通運算子。IEEE 影像處理交易,7(4),555-570。DOI:10.1109/83.663500
[4]Najman, L., & Couprie, M. (2006)。以擬線性時間建立元件樹。IEEE 影像處理交易,15(11),3531-3539。DOI:10.1109/TIP.2006.877518
[5]Carlinet, E., & Geraud, T. (2014)。元件樹計算演算法的比較評論。IEEE 影像處理交易,23(9),3885-3895。DOI:10.1109/TIP.2014.2336551
範例
我們建立一個影像(中心具有最大值的二次函數,以及 4 個額外的局部最大值)。
>>> w = 12 >>> x, y = np.mgrid[0:w,0:w] >>> f = 20 - 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:3,1:5] = 40; f[2:4,9:11] = 60; f[9:11,2:4] = 80 >>> f[9:10,9:11] = 100; f[10,10] = 100 >>> f = f.astype(int)
我們可以計算面積開運算。
>>> open = area_opening(f, 8, connectivity=1)
移除表面積小於 8 的峰值。
- skimage.morphology.ball(radius, dtype=<class 'numpy.uint8'>, *, strict_radius=True, decomposition=None)[來源]#
產生球狀的足跡。
這是圓盤的 3D 等效物。如果像素與原點之間的歐幾里得距離不大於半徑,則該像素位於鄰域內。
- 參數:
- radiusfloat
球形足跡的半徑。
- 回傳:
- footprintndarray 或 tuple
足跡,其中鄰域的元素為 1,否則為 0。
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- strict_radiusbool,可選
如果為 False,則將半徑擴展 0.5。這允許圓在沿每個軸保持大小
2 * radius + 1的立方體內進一步擴展。如果 decomposition 不是 None,則忽略此參數。- decomposition{None, 'sequence'},可選
如果為 None,則返回單個陣列。對於 'sequence',則返回較小足跡的元組。應用這一系列較小的足跡將產生與單個較大足跡等效的結果,但具有更好的計算效能。對於球形足跡,序列分解與 decomposition=None 不完全等效。請參閱注意事項以獲取更多詳細資訊。
注意
由 decomposition='sequence' 模式產生的圓盤與 decomposition=None 的圓盤不相同。在這裡,我們將 [1] 中針對圓盤所採用的方法擴展到 3D 情況,使用該出版物中「正方形」、「菱形」和「T 形」元素的 3 維擴展。所有這些基本元素的大小都是
(3,) * ndim。我們以數值方式計算每個元素的重複次數,以最接近使用 kwargsstrict_radius=False, decomposition=None計算的球體。根據經驗,序列分解的等效複合足跡接近菱形截角八面體 (26 面 [2])。
參考文獻
[1]Park, H and Chin R.T. Decomposition of structuring elements for optimal implementation of morphological operations. In Proceedings: 1997 IEEE Workshop on Nonlinear Signal and Image Processing, London, UK. https://www.iwaenc.org/proceedings/1997/nsip97/pdf/scan/ns970226.pdf
- skimage.morphology.binary_closing(image, footprint=None, out=None, *, mode='ignore')[來源]#
回傳影像的快速二元形態學閉合。
此函數返回與灰階閉運算相同的結果,但對於二值影像執行速度更快。
影像的形態閉運算定義為膨脹後接侵蝕。閉運算可以去除小的暗點(即「胡椒」)並連接小的明亮裂縫。這傾向於「封閉」特徵之間(明亮)的(黑暗)間隙。
- 參數:
- imagendarray
二值輸入影像。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效模式為:'max'、'min'、'ignore'。如果為 'ignore',則假設影像域外的像素對於侵蝕為True,對於膨脹為False,這會導致它們不影響結果。預設值為 'ignore'。在 0.23 版本中新增:
mode在 0.23 中新增。
- 回傳:
- closingbool 的 ndarray
形態閉運算的結果。
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。
- skimage.morphology.binary_dilation(image, footprint=None, out=None, *, mode='ignore')[來源]#
回傳影像的快速二元形態學膨脹。
此函數返回與灰階膨脹相同的結果,但對於二值影像執行速度更快。
形態學膨脹將
(i,j)處的像素設定為以(i,j)為中心的鄰域中所有像素的最大值。膨脹會擴大明亮區域並縮小暗區域。- 參數:
- imagendarray
二值輸入影像。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效模式為:'max'、'min'、'ignore'。如果為 'min' 或 'ignore',則假設影像域外的像素為False,這會導致它們不影響結果。預設值為 'ignore'。在 0.23 版本中新增:
mode在 0.23 中新增。
- 回傳:
- dilatedbool 或 uint 的 ndarray
形態學膨脹的結果,值在
[False, True]中。
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。對於非對稱足跡,
skimage.morphology.binary_dilation()和skimage.morphology.dilation()產生的輸出不同:binary_dilation反射足跡,而dilation不反射。
- skimage.morphology.binary_erosion(image, footprint=None, out=None, *, mode='ignore')[原始碼]#
回傳影像的快速二元形態學侵蝕。
此函數返回與灰度侵蝕相同的結果,但對於二元影像執行速度更快。
形態學侵蝕將
(i,j)位置的像素設定為以(i,j)為中心的所有鄰域像素的最小值。侵蝕會縮小明亮區域並擴大黑暗區域。- 參數:
- imagendarray
二值輸入影像。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效的模式為:‘max’、‘min’、‘ignore’。如果為 ‘max’ 或 ‘ignore’,則假設影像範圍外的像素為True,這會導致它們不影響結果。預設值為 ‘ignore’。在 0.23 版本中新增:
mode在 0.23 中新增。
- 回傳:
- eroded布林或 uint 的 ndarray
形態學侵蝕的結果,其值在
[False, True]之間。
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。對於偶數尺寸的足跡,
skimage.morphology.erosion()和此函數產生的輸出會有所不同:一個相對於另一個移動一個像素。
- skimage.morphology.binary_opening(image, footprint=None, out=None, *, mode='ignore')[原始碼]#
回傳影像的快速二元形態學開啟。
此函數返回與灰度開運算相同的結果,但對於二元影像執行速度更快。
影像的形態學開運算定義為侵蝕後再進行膨脹。開運算可以去除小的明亮點(即「鹽」)並連接小的黑暗裂縫。這往往會「打開」(黑暗的)特徵之間的間隙。
- 參數:
- imagendarray
二值輸入影像。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效模式為:'max'、'min'、'ignore'。如果為 'ignore',則假設影像域外的像素對於侵蝕為True,對於膨脹為False,這會導致它們不影響結果。預設值為 'ignore'。在 0.23 版本中新增:
mode在 0.23 中新增。
- 回傳:
- opening布林的 ndarray
形態學開運算的結果。
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。
- skimage.morphology.black_tophat(image, footprint=None, out=None, *, mode='reflect', cval=0.0)[原始碼]#
回傳影像的黑色禮帽。
影像的黑色禮帽定義為其形態學閉運算減去原始影像。此運算返回影像中比足跡小的黑暗點。請注意,原始影像中的黑暗點在黑色禮帽之後會變成亮點。
- 參數:
- imagendarray
影像陣列。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outndarray,選用
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效的模式為:‘reflect’、‘constant’、‘nearest’、‘mirror’、‘wrap’、‘max’、‘min’ 或 ‘ignore’。請參閱skimage.morphology.closing()。預設值為 ‘reflect’。- cval純量,選用
如果
mode為 ‘constant’,則填充輸入邊緣外的值。預設值為 0.0。在 0.23 版中新增:
mode和cval是在 0.23 版中新增的。
- 回傳:
- out陣列,與
image具有相同的形狀和類型 形態學黑色禮帽的結果。
- out陣列,與
另請參閱
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。參考文獻
範例
>>> # Change dark peak to bright peak and subtract background >>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> dark_on_gray = np.array([[7, 6, 6, 6, 7], ... [6, 5, 4, 5, 6], ... [6, 4, 0, 4, 6], ... [6, 5, 4, 5, 6], ... [7, 6, 6, 6, 7]], dtype=np.uint8) >>> black_tophat(dark_on_gray, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [0, 0, 1, 0, 0], [0, 1, 5, 1, 0], [0, 0, 1, 0, 0], [0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.closing(image, footprint=None, out=None, *, mode='reflect', cval=0.0)[原始碼]#
回傳影像的灰階形態學閉合。
影像的形態學閉運算定義為膨脹後再進行侵蝕。閉運算可以去除小的黑暗點(即「胡椒」)並連接小的明亮裂縫。這往往會「關閉」(黑暗的)特徵之間的間隙。
- 參數:
- imagendarray
影像陣列。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outndarray,選用
用於儲存形態學結果的陣列。如果為 None,則將會分配一個新的陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效的模式為:‘reflect’、‘constant’、‘nearest’、‘mirror’、‘wrap’、‘max’、‘min’ 或 ‘ignore’。如果為 ‘ignore’,則假設影像範圍外的像素在侵蝕中為影像 dtype 的最大值,而在膨脹中則為最小值,這會導致它們不影響結果。預設值為 ‘reflect’。- cval純量,選用
如果
mode為 ‘constant’,則填充輸入邊緣外的值。預設值為 0.0。在 0.23 版中新增:
mode和cval是在 0.23 版中新增的。
- 回傳:
- closing陣列,與
image具有相同的形狀和類型 形態閉運算的結果。
- closing陣列,與
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。範例
>>> # Close a gap between two bright lines >>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> broken_line = np.array([[0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0], ... [1, 1, 0, 1, 1], ... [0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint8) >>> closing(broken_line, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [0, 0, 0, 0, 0], [1, 1, 1, 1, 1], [0, 0, 0, 0, 0], [0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.convex_hull_image(image, offset_coordinates=True, tolerance=1e-10, include_borders=True)[原始碼]#
計算二元影像的凸包影像。
凸包是包含在最小凸多邊形中,環繞輸入影像中所有白色像素的像素集合。
- 參數:
- image陣列
二元輸入影像。此陣列在處理前會轉換為布林值。
- offset_coordinates布林值,選用
如果
True,則座標處的像素(例如 (4, 7))將由座標 (3.5, 7)、(4.5, 7)、(4, 6.5) 和 (4, 7.5) 表示。這在計算外殼時會為像素增加一些「範圍」。- tolerance浮點數,選用
在判斷某個點是否在凸包內時的容差。由於數值浮點錯誤,容差為 0 會導致某些點錯誤地分類為在凸包外。
- include_borders:布林值,選用
如果
False,則最終凸包遮罩中將會排除頂點/邊緣。
- 回傳:
- hull布林的 (M, N) 陣列
二元影像,其中凸包中的像素設定為 True。
參考文獻
- skimage.morphology.convex_hull_object(image, *, connectivity=2)[原始碼]#
計算二元影像中個別物件的凸包影像。
凸包是包含在最小凸多邊形中,環繞輸入影像中所有白色像素的像素集合。
- 參數:
- image(M, N) ndarray
二值輸入影像。
- connectivity{1, 2}, int, 選用
決定每個像素的鄰居。從像素中心開始,在
connectivity的平方距離內的相鄰元素會被視為鄰居。1-connectivity 2-connectivity [ ] [ ] [ ] [ ] | \ | / [ ]--[x]--[ ] [ ]--[x]--[ ] | / | \ [ ] [ ] [ ] [ ]
- 回傳:
- hull布林的 ndarray
二元影像,其中凸包內的像素設定為
True。
注意
此函數使用
skimage.morphology.label來定義獨立的物件,使用convex_hull_image找出每個物件的凸包,並以邏輯 OR 運算合併這些區域。請注意,不相連物件的凸包可能會在結果中重疊。如果懷疑會發生這種情況,請考慮對每個物件分別使用 convex_hull_image,或調整connectivity。
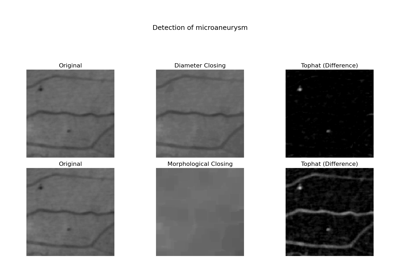
- skimage.morphology.diameter_closing(image, diameter_threshold=8, connectivity=1, parent=None, tree_traverser=None)[原始碼]#
執行影像的直徑閉合。
直徑閉合會移除影像中最大延伸小於 diameter_threshold 的所有暗結構。最大延伸定義為邊界框的最大延伸。此運算子也稱為邊界框閉合。實際上,結果類似於形態學閉合,但不會移除長而細的結構。
從技術上講,此運算子基於影像的最大樹表示。
- 參數:
- imagendarray
要計算直徑閉合的輸入影像。此影像可以是任何類型。
- diameter_threshold無號整數
最大延伸參數 (像素數)。預設值為 8。
- connectivity無號整數,選用
鄰域連通性。整數表示到達鄰居的最大正交步數。在 2D 中,4 鄰域為 1,而 8 鄰域為 2。預設值為 1。
- parentndarray, int64,選用
預先計算的父影像,代表反轉影像的最大樹狀結構。如果提供預先計算的 parent 和 tree_traverser,此函數會很快。請參閱「注意事項」以取得更多詳細資訊。
- tree_traverser1D 陣列,int64,選用
預先計算的遍歷器,其中像素的順序為每個像素在其父像素之前(根像素沒有父像素)。如果提供預先計算的 parent 和 tree_traverser,此函數會很快。請參閱「注意事項」以取得更多詳細資訊。
- 回傳:
- outputndarray
輸出影像的形狀和類型與輸入影像相同。
另請參閱
注意
如果將最大樹表示法(父項和 tree_traverser)提供給函式,則必須從此函式的反轉影像計算它們,即:>>> P, S = max_tree(invert(f)) >>> closed = diameter_closing(f, 3, parent=P, tree_traverser=S)
參考文獻
[1]Walter, T., & Klein, J.-C. (2002). Automatic Detection of Microaneurysms in Color Fundus Images of the Human Retina by Means of the Bounding Box Closing. In A. Colosimo, P. Sirabella, A. Giuliani (Eds.), Medical Data Analysis. Lecture Notes in Computer Science, vol 2526, pp. 210-220. Springer Berlin Heidelberg. DOI:10.1007/3-540-36104-9_23
[2]Carlinet, E., & Geraud, T. (2014)。元件樹計算演算法的比較評論。IEEE 影像處理交易,23(9),3885-3895。DOI:10.1109/TIP.2014.2336551
範例
我們建立一個影像(在中心具有最小值和 4 個額外局部最小值的二次函數)。
>>> w = 12 >>> x, y = np.mgrid[0:w,0:w] >>> f = 180 + 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:3,1:5] = 160; f[2:4,9:11] = 140; f[9:11,2:4] = 120 >>> f[9:10,9:11] = 100; f[10,10] = 100 >>> f = f.astype(int)
我們可以計算直徑閉合
>>> closed = diameter_closing(f, 3, connectivity=1)
移除所有最大延伸為 2 或更小的所有小最小值。剩餘的最小值都具有至少 3 的最大延伸。
- skimage.morphology.diameter_opening(image, diameter_threshold=8, connectivity=1, parent=None, tree_traverser=None)[原始碼]#
執行影像的直徑開啟。
直徑開口會移除影像中最大延伸小於 diameter_threshold 的所有亮結構。最大延伸定義為邊界框的最大延伸。此運算子也稱為邊界框開口。實際上,結果類似於形態學開口,但不會移除長而細的結構。
從技術上講,此運算子基於影像的最大樹表示。
- 參數:
- imagendarray
要計算面積開啟的輸入影像。此影像可以是任何類型。
- diameter_threshold無號整數
最大延伸參數 (像素數)。預設值為 8。
- connectivity無號整數,選用
鄰域連通性。整數表示到達鄰居的最大正交步數。在 2D 中,4 鄰域為 1,而 8 鄰域為 2。預設值為 1。
- parentndarray, int64,選用
父圖像,表示該影像的最大樹。每個像素的值是其在展開陣列中父節點的索引。
- tree_traverser1D 陣列,int64,選用
已排序的像素索引(參考展開的陣列)。像素的排序方式為每個像素都位於其父項之前(根除外,根沒有父項)。
- 回傳:
- outputndarray
輸出影像,與輸入影像具有相同的形狀和類型。
另請參閱
參考文獻
[1]Walter, T., & Klein, J.-C. (2002). Automatic Detection of Microaneurysms in Color Fundus Images of the Human Retina by Means of the Bounding Box Closing. In A. Colosimo, P. Sirabella, A. Giuliani (Eds.), Medical Data Analysis. Lecture Notes in Computer Science, vol 2526, pp. 210-220. Springer Berlin Heidelberg. DOI:10.1007/3-540-36104-9_23
[2]Carlinet, E., & Geraud, T. (2014)。元件樹計算演算法的比較評論。IEEE 影像處理交易,23(9),3885-3895。DOI:10.1109/TIP.2014.2336551
範例
我們建立一個影像(中心具有最大值的二次函數,以及 4 個額外的局部最大值)。
>>> w = 12 >>> x, y = np.mgrid[0:w,0:w] >>> f = 20 - 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:3,1:5] = 40; f[2:4,9:11] = 60; f[9:11,2:4] = 80 >>> f[9:10,9:11] = 100; f[10,10] = 100 >>> f = f.astype(int)
我們可以計算直徑開口
>>> open = diameter_opening(f, 3, connectivity=1)
移除最大延伸為 2 或更小的峰值。剩餘的峰值都具有至少 3 的最大延伸。
- skimage.morphology.diamond(radius, dtype=<class 'numpy.uint8'>, *, decomposition=None)[原始碼]#
產生平坦的菱形足跡。
如果像素與鄰域中心之間的城市區塊/曼哈頓距離不超過半徑,則該像素是鄰域的一部分(即標記為 1)。
- 參數:
- radius整數
菱形形狀足跡的半徑。
- 回傳:
- footprintndarray 或 tuple
足跡,其中鄰域的元素為 1,否則為 0。當
decomposition為 None 時,這只是一個 numpy.ndarray。否則,這會是一個 tuple,其長度等於要套用的唯一結構元素的數量(請參閱「注意事項」以取得更多詳細資訊)
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- decomposition{None, 'sequence'},可選
如果為 None,則會傳回單一陣列。對於 ‘sequence’,會傳回較小足跡的 tuple。套用這一系列的較小足跡,會得到與單一較大足跡相同的結果,但具有更好的計算效能。請參閱「注意事項」以取得更多詳細資訊。
注意
當
decomposition不為 None 時,footprinttuple 的每個元素都是(ndarray, num_iter)形式的 2-tuple,指定足跡陣列及其要套用的疊代次數。對於二元或灰階形態學,觀察到使用
decomposition='sequence'具有效能優勢,且優勢的大小隨著足跡大小的增加而增加。
- skimage.morphology.dilation(image, footprint=None, out=None, shift_x=<DEPRECATED>, shift_y=<DEPRECATED>, *, mode='reflect', cval=0.0)[原始碼]#
回傳影像的灰階形態學膨脹。
形態學膨脹會將像素的值設定為以其為中心的局部鄰域內的所有像素值的最大值。足跡為 1 的值定義此鄰域。膨脹會放大亮區域並縮小暗區域。
- 參數:
- imagendarray
影像陣列。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outndarray,選用
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效的模式為:「reflect」、「constant」、「nearest」、「mirror」、「wrap」、「max」、「min」或「ignore」。如果是「min」或「ignore」,則假設影像域之外的像素是影像 dtype 的最大值,這會導致它們不影響結果。預設值為「reflect」。- cval純量,選用
如果
mode為 ‘constant’,則填充輸入邊緣外的值。預設值為 0.0。在 0.23 版中新增:
mode和cval是在 0.23 版中新增的。
- 回傳:
- dilateduint8 陣列,形狀和類型與
image相同 形態學膨脹的結果。
- dilateduint8 陣列,形狀和類型與
- 其他參數:
- shift_x、shift_y已棄用
自版本 0.23 起已棄用。
注意
對於
uint8(和uint16直到某個位元深度)資料,較低的演算法複雜度使得skimage.filters.rank.maximum()函數對於較大的影像和足跡更有效率。足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。對於非對稱足跡,
skimage.morphology.binary_dilation()和skimage.morphology.dilation()產生的輸出不同:binary_dilation反射足跡,而dilation不反射。範例
>>> # Dilation enlarges bright regions >>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> bright_pixel = np.array([[0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0], ... [0, 0, 1, 0, 0], ... [0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint8) >>> dilation(bright_pixel, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [0, 1, 1, 1, 0], [0, 1, 1, 1, 0], [0, 1, 1, 1, 0], [0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.disk(radius, dtype=<class 'numpy.uint8'>, *, strict_radius=True, decomposition=None)[原始碼]#
產生平坦的圓盤狀足跡。
如果像素與原點之間的歐幾里得距離不超過半徑,則該像素位於鄰域內(僅當
decomposition == 'sequence'時,這僅為近似值)。- 參數:
- radius整數
圓盤形狀足跡的半徑。
- 回傳:
- footprintndarray
足跡,其中鄰域的元素為 1,否則為 0。
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- strict_radiusbool,可選
如果為 False,則將半徑擴展 0.5。這允許圓在沿每個軸保持大小
2 * radius + 1的立方體內進一步擴展。如果 decomposition 不是 None,則忽略此參數。- decomposition{None, ‘sequence’, ‘crosses’}, 選用
如果為 None,則會傳回單一陣列。對於 ‘sequence’,會傳回較小足跡的 tuple。套用這一系列的較小足跡,會得到與單一較大足跡等效的結果,但具有更好的計算效能。對於圓盤足跡,‘sequence’ 或 ‘crosses’ 分解並不總是與
decomposition=None完全等效。請參閱「注意事項」以取得更多詳細資訊。
注意
當
decomposition不為 None 時,footprinttuple 的每個元素都是(ndarray, num_iter)形式的 2-tuple,指定足跡陣列及其要套用的疊代次數。使用
decomposition='sequence'模式產生的圓盤可能與使用decomposition=None產生的圓盤不完全相同。圓盤的覆蓋區可以通過沿每個軸應用一系列範圍為 3 的較小覆蓋區來近似。有關此的具體解決方案在 [1] 中給出,適用於半徑為 2 到 10 的 2D 圓盤。在這裡,我們數值計算了每個元素的重複次數,以最接近使用 kwargsstrict_radius=False, decomposition=None計算出的圓盤。根據經驗,大半徑下的序列分解會接近十六邊形(一個 16 邊的多邊形 [2])。在 [3] 中,作者證明對於使用形狀為 (3, 3) 的覆蓋區進行分解,十六邊形是圓盤最接近的近似值。
使用
decomposition='crosses'產生的圓盤通常(但並非總是)與使用decomposition=None產生的圓盤相同。它往往比decomposition='sequence'給出更接近的近似值,且效能相當。個別十字形元素的尺寸不限於 (3, 3)。與 'sequence' 分解不同,'crosses' 分解也可以精確地近似於具有strict_radius=True的圓盤形狀。該方法基於對 [4] 中給出的演算法 1 的改編。參考文獻
[1]Park, H and Chin R.T. Decomposition of structuring elements for optimal implementation of morphological operations. In Proceedings: 1997 IEEE Workshop on Nonlinear Signal and Image Processing, London, UK. https://www.iwaenc.org/proceedings/1997/nsip97/pdf/scan/ns970226.pdf
[3]Vanrell, M and Vitrià, J. Optimal 3 × 3 decomposable disks for morphological transformations. Image and Vision Computing, Vol. 15, Issue 11, 1997. DOI:10.1016/S0262-8856(97)00026-7
[4]Li, D. and Ritter, G.X. Decomposition of Separable and Symmetric Convex Templates. Proc. SPIE 1350, Image Algebra and Morphological Image Processing, (1 November 1990). DOI:10.1117/12.23608
- skimage.morphology.ellipse(width, height, dtype=<class 'numpy.uint8'>, *, decomposition=None)[來源]#
產生平坦的橢圓形足跡。
橢圓周邊的每個像素都滿足方程式
(x/width+1)**2 + (y/height+1)**2 = 1。- 參數:
- width整數
橢圓形覆蓋區的寬度。
- height整數
橢圓形覆蓋區的高度。
- 回傳:
- footprintndarray
鄰域元素為 1,否則為 0 的覆蓋區。覆蓋區的形狀將為
(2 * height + 1, 2 * width + 1)。
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- decomposition{None, 'crosses'}, 選項
如果為 None,則會傳回單一陣列。對於 ‘sequence’,會傳回較小足跡的 tuple。套用這一系列的較小足跡,會得到與單一較大足跡相同的結果,但具有更好的計算效能。請參閱「注意事項」以取得更多詳細資訊。
注意
當
decomposition不為 None 時,footprinttuple 的每個元素都是(ndarray, num_iter)形式的 2-tuple,指定足跡陣列及其要套用的疊代次數。使用
decomposition='crosses'產生的橢圓通常(但並非總是)與使用decomposition=None產生的橢圓相同。該方法基於對 [1] 中給出的演算法 1 的改編。參考文獻
[1]Li, D. and Ritter, G.X. Decomposition of Separable and Symmetric Convex Templates. Proc. SPIE 1350, Image Algebra and Morphological Image Processing, (1 November 1990). DOI:10.1117/12.23608
範例
>>> from skimage.morphology import footprints >>> footprints.ellipse(5, 3) array([[0, 0, 1, 1, 1, 1, 1, 1, 1, 0, 0], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [0, 0, 1, 1, 1, 1, 1, 1, 1, 0, 0]], dtype=uint8)
- skimage.morphology.erosion(image, footprint=None, out=None, shift_x=<DEPRECATED>, shift_y=<DEPRECATED>, *, mode='reflect', cval=0.0)[來源]#
回傳影像的灰階形態學侵蝕。
形態侵蝕將 (i,j) 處的像素設置為以 (i,j) 為中心的所有鄰域像素中的最小值。侵蝕會縮小亮區並擴大暗區。
- 參數:
- imagendarray
影像陣列。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outndarrays,選項
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效模式包括:'reflect'、'constant'、'nearest'、'mirror'、'wrap'、'max'、'min' 或 'ignore'。如果為 'max' 或 'ignore',則假設圖像域外的像素為圖像 dtype 的最大值,這會導致它們不影響結果。預設值為 'reflect'。- cval純量,選用
如果
mode為 ‘constant’,則填充輸入邊緣外的值。預設值為 0.0。在 0.23 版中新增:
mode和cval是在 0.23 版中新增的。
- 回傳:
- eroded陣列,形狀與
image相同 形態侵蝕的結果。
- eroded陣列,形狀與
- 其他參數:
- shift_x、shift_y已棄用
自版本 0.23 起已棄用。
注意
對於
uint8(以及直到特定位元深度的uint16)資料,較低的演算法複雜度使得skimage.filters.rank.minimum()函數對於較大的圖像和覆蓋區更有效。足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。對於偶數大小的覆蓋區,
skimage.morphology.binary_erosion()和此函數產生的輸出有所不同:一個輸出與另一個輸出相比會偏移一個像素。範例
>>> # Erosion shrinks bright regions >>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> bright_square = np.array([[0, 0, 0, 0, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 1, 1, 1, 0], ... [0, 0, 0, 0, 0]], dtype=np.uint8) >>> erosion(bright_square, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [0, 0, 0, 0, 0], [0, 0, 1, 0, 0], [0, 0, 0, 0, 0], [0, 0, 0, 0, 0]], dtype=uint8)
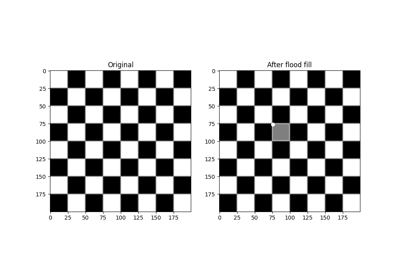
- skimage.morphology.flood(image, seed_point, *, footprint=None, connectivity=None, tolerance=None)[來源]#
對應到洪水填充的遮罩。
從特定的
seed_point開始,會找到與種子值相等或在tolerance範圍內的連線點。- 參數:
- imagendarray
一個 n 維陣列。
- seed_point元組或整數
image中用作洪水填充起點的點。如果影像為 1D,則此點可表示為整數。- footprintndarray,選項
用於確定每個評估像素的鄰域的覆蓋區(結構元素)。它必須僅包含 1 和 0,並且具有與
image相同的維度數。如果未給定,則會將所有相鄰像素視為鄰域的一部分(完全連線)。- connectivity整數,選項
一個用於確定每個評估像素的鄰域的數字。與中心平方距離小於或等於
connectivity的相鄰像素被視為鄰居。如果footprint不是 None,則會忽略。- tolerance浮點數或整數,選項
如果為 None(預設值),則相鄰值必須與
image在seed_point的初始值完全相等。這是最快的。如果給定一個值,則會在每個點進行比較,如果該值在初始值的容差範圍內,也會被填充(含)。
- 回傳:
- maskndarray
會傳回與
image形狀相同的布林值陣列,其中 True 值表示與種子點連線並等於(或在容差範圍內)的區域。所有其他值均為 False。
注意
此運算的觀念類比是許多點陣圖形程式中的「油漆桶」工具。此函數僅傳回表示填充的遮罩。
如果為了節省記憶體而需要索引而不是遮罩,使用者可以簡單地在結果上執行
numpy.nonzero、儲存索引並捨棄此遮罩。範例
>>> from skimage.morphology import flood >>> image = np.zeros((4, 7), dtype=int) >>> image[1:3, 1:3] = 1 >>> image[3, 0] = 1 >>> image[1:3, 4:6] = 2 >>> image[3, 6] = 3 >>> image array([[0, 0, 0, 0, 0, 0, 0], [0, 1, 1, 0, 2, 2, 0], [0, 1, 1, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
使用完整連線性(包括對角線)將連線的 1 填充為 5
>>> mask = flood(image, (1, 1)) >>> image_flooded = image.copy() >>> image_flooded[mask] = 5 >>> image_flooded array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [5, 0, 0, 0, 0, 0, 3]])
將連線的 1 填充為 5,排除對角線點(連線性 1)
>>> mask = flood(image, (1, 1), connectivity=1) >>> image_flooded = image.copy() >>> image_flooded[mask] = 5 >>> image_flooded array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
使用容差填充
>>> mask = flood(image, (0, 0), tolerance=1) >>> image_flooded = image.copy() >>> image_flooded[mask] = 5 >>> image_flooded array([[5, 5, 5, 5, 5, 5, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 5, 5, 3]])
- skimage.morphology.flood_fill(image, seed_point, new_value, *, footprint=None, connectivity=None, tolerance=None, in_place=False)[來源]#
在影像上執行洪水填充。
從特定的
seed_point開始,尋找與種子值相等或在tolerance範圍內的連通點,然後將這些點設定為new_value。- 參數:
- imagendarray
一個 n 維陣列。
- seed_point元組或整數
image中用作洪水填充起點的點。如果影像為 1D,則此點可表示為整數。- new_value
image類型 要設定整個填充區域的新值。此值必須與
image的 dtype 一致。- footprintndarray,選項
用於確定每個評估像素的鄰域的覆蓋區(結構元素)。它必須僅包含 1 和 0,並且具有與
image相同的維度數。如果未給定,則會將所有相鄰像素視為鄰域的一部分(完全連線)。- connectivity整數,選項
一個用於確定每個評估像素的鄰域的數字。與中心平方距離小於或等於
connectivity的相鄰像素被視為鄰居。如果footprint不是 None,則會忽略。- tolerance浮點數或整數,選項
如果為 None (預設值),則相鄰值必須與
image在seed_point的值完全相等,才能進行填充。這是最快的方式。如果提供了容差值,則會填充值在種子點值正負容差範圍內的相鄰點(包含邊界值)。- in_placebool,可選
如果為 True,則原地對
image應用洪水填充。如果為 False,則返回洪水填充的結果,而不修改輸入的image(預設值)。
- 回傳:
- filledndarray
返回一個形狀與
image相同的陣列,其中與種子點連通且相等 (或在容差範圍內) 的區域中的值會被替換為new_value。
注意
此操作的概念類比是許多點陣圖形程式中的「油漆桶」工具。
範例
>>> from skimage.morphology import flood_fill >>> image = np.zeros((4, 7), dtype=int) >>> image[1:3, 1:3] = 1 >>> image[3, 0] = 1 >>> image[1:3, 4:6] = 2 >>> image[3, 6] = 3 >>> image array([[0, 0, 0, 0, 0, 0, 0], [0, 1, 1, 0, 2, 2, 0], [0, 1, 1, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
使用完整連線性(包括對角線)將連線的 1 填充為 5
>>> flood_fill(image, (1, 1), 5) array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [5, 0, 0, 0, 0, 0, 3]])
將連線的 1 填充為 5,排除對角線點(連線性 1)
>>> flood_fill(image, (1, 1), 5, connectivity=1) array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
使用容差填充
>>> flood_fill(image, (0, 0), 5, tolerance=1) array([[5, 5, 5, 5, 5, 5, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 5, 5, 3]])
- skimage.morphology.footprint_from_sequence(footprints)[來源]#
將足跡序列轉換為等效的 ndarray。
- 參數:
- footprints2 元組的元組
足跡元組的序列,其中每個元組的第一個元素是對應於足跡的陣列,第二個元素是要應用該足跡的次數。目前,所有足跡的大小都應該是奇數。
- 回傳:
- footprintndarray
與應用
footprints序列等效的單個陣列。
- skimage.morphology.footprint_rectangle(shape, *, dtype=<class 'numpy.uint8'>, decomposition=None)[來源]#
產生矩形或超矩形足跡。
根據使用
shape請求的長度和維度,產生正方形、矩形、立方體、長方體,甚至是這些形狀的更高維度版本。- 參數:
- shapetuple[int, …]
每個維度中足跡的長度。序列的長度決定足跡的維度數量。
- dtype資料類型,可選
足跡的資料類型。
- decomposition{None, ‘separable’, ‘sequence’}, 可選
如果為 None,則返回單個陣列。對於 ‘sequence’,則返回較小足跡的元組。應用這一系列的較小足跡將產生與單個較大足跡相同的結果,但通常具有更好的計算效能。有關更多詳細資訊,請參閱注意事項。對於 ‘separable’,此函數會針對每個軸使用可分離的 1D 足跡。’sequence’ 或 ‘separable’ 在計算上哪一個更快,可能取決於架構。
- 回傳:
- footprint陣列或 tuple[tuple[ndarray, int], …]
一個僅由 1 組成的足跡,也就是說,每個像素都屬於鄰域。當
decomposition為 None 時,這只是一個陣列。否則,這將是一個元組,其長度等於要應用之唯一結構元素的數量 (請參閱範例了解更多詳細資訊)。
範例
>>> import skimage as ski >>> ski.morphology.footprint_rectangle((3, 5)) array([[1, 1, 1, 1, 1], [1, 1, 1, 1, 1], [1, 1, 1, 1, 1]], dtype=uint8)
分解將傳回多個足跡,這些足跡組合成所請求形狀的簡單足跡。
>>> ski.morphology.footprint_rectangle((9, 9), decomposition="sequence") ((array([[1, 1, 1], [1, 1, 1], [1, 1, 1]], dtype=uint8), 4),)
"sequence"確保分解只返回 1D 足跡。>>> ski.morphology.footprint_rectangle((3, 5), decomposition="separable") ((array([[1], [1], [1]], dtype=uint8), 1), (array([[1, 1, 1, 1, 1]], dtype=uint8), 1))
產生一個 5 維超立方體,每個維度有 3 個樣本
>>> ski.morphology.footprint_rectangle((3,) * 5).shape (3, 3, 3, 3, 3)
- skimage.morphology.h_maxima(image, h, footprint=None)[來源]#
決定影像中所有高度 >= h 的最大值。
局部最大值定義為連通的像素集合,其灰階值嚴格大於該集合直接鄰域中所有像素的灰階值。
高度為 h 的局部最大值 M 是一個局部最大值,它至少有一條路徑連接 M 與相等或更高的局部最大值,該路徑上的最小值為 f(M) - h(即,路徑上的值相對於最大值下降不超過 h),並且沒有任何路徑連接到相等或更高的局部最大值,其最小值更大。
此函數也會找到影像的全域最大值。
- 參數:
- imagendarray
要計算最大值的輸入影像。
- h無號整數
所有提取的最大值的最小高度。
- footprintndarray,選項
鄰域表示為 1 和 0 的 n 維陣列。預設值是根據最大範數的半徑為 1 的球體(即,對於 2D 影像,為 3x3 正方形;對於 3D 影像,為 3x3x3 立方體等)。
- 回傳:
- h_maxndarray
高度 >= h 的局部最大值和全域最大值。結果影像是一個二元影像,其中屬於已確定最大值的像素值為 1,其他像素的值為 0。
參考文獻
[1]Soille, P.,“形態影像分析:原理與應用”(第 6 章),第二版 (2003),ISBN 3540429883。
範例
>>> import numpy as np >>> from skimage.morphology import extrema
我們建立一個影像(中心有最大值的二次函數,以及 4 個額外的常數最大值。最大值的高度為:1、21、41、61、81
>>> w = 10 >>> x, y = np.mgrid[0:w,0:w] >>> f = 20 - 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:4,2:4] = 40; f[2:4,7:9] = 60; f[7:9,2:4] = 80; f[7:9,7:9] = 100 >>> f = f.astype(int)
我們可以計算高度至少為 40 的所有最大值
>>> maxima = extrema.h_maxima(f, 40)
結果影像將包含 3 個局部最大值。
- skimage.morphology.h_minima(image, h, footprint=None)[來源]#
決定影像中所有深度 >= h 的最小值。
局部最小值定義為連通的像素集合,其灰階值嚴格小於該集合直接鄰域中所有像素的灰階值。
深度為 h 的局部最小值 M 是一個局部最小值,它至少有一條路徑連接 M 與相等或更低的局部最小值,該路徑上的最大值為 f(M) + h(即,路徑上的值相對於最小值增加不超過 h),並且沒有任何路徑連接到相等或更低的局部最小值,其最大值更小。
此函數也會找到影像的全域最小值。
- 參數:
- imagendarray
要計算最小值的輸入影像。
- h無號整數
所有提取的最小值的最小深度。
- footprintndarray,選項
鄰域表示為 1 和 0 的 n 維陣列。預設值是根據最大範數的半徑為 1 的球體(即,對於 2D 影像,為 3x3 正方形;對於 3D 影像,為 3x3x3 立方體等)。
- 回傳:
- h_minndarray
深度 >= h 的局部最小值和全域最小值。結果影像是一個二元影像,其中屬於已確定最小值的像素值為 1,其他像素的值為 0。
參考文獻
[1]Soille, P.,“形態影像分析:原理與應用”(第 6 章),第二版 (2003),ISBN 3540429883。
範例
>>> import numpy as np >>> from skimage.morphology import extrema
我們建立一個影像(中心有最小值的二次函數,以及 4 個額外的常數最大值。最小值的深度為:1、21、41、61、81
>>> w = 10 >>> x, y = np.mgrid[0:w,0:w] >>> f = 180 + 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:4,2:4] = 160; f[2:4,7:9] = 140; f[7:9,2:4] = 120; f[7:9,7:9] = 100 >>> f = f.astype(int)
我們可以計算深度至少為 40 的所有最小值
>>> minima = extrema.h_minima(f, 40)
結果影像將包含 3 個局部最小值。
- skimage.morphology.isotropic_closing(image, radius, out=None, spacing=None)[來源]#
回傳影像的二元形態學閉合。
此函數返回與二元
skimage.morphology.binary_closing()相同的結果,但對於較大的圓形結構元素,執行速度更快。這是透過對精確歐幾里得距離圖 [1]、[2] 進行閾值處理來實現的。此實作基於:func:scipy.ndimage.distance_transform_edt。- 參數:
- imagendarray
二值輸入影像。
- radiusfloat
應關閉區域的半徑。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- spacingfloat 或 float 序列,可選
沿每個維度元素的間距。如果為序列,則長度必須等於輸入的維度(軸數)。如果為單個數字,則此值會用於所有軸。如果未指定,則表示格點間距為 1。
- 回傳:
- closedbool 的 ndarray
形態閉運算的結果。
參考文獻
[1]Cuisenaire, O. 和 Macq, B.,“使用透過傳播的局部距離轉換的快速歐幾里得形態運算子及其應用”,1999 年影像處理及其應用,第七屆國際會議(會議出版物第 465 號),1999 年,第 856-860 頁,第 2 卷。DOI:10.1049/cp:19990446
[2]Ingemar Ragnemalm, 透過輪廓處理和距離圖的閾值處理實現的快速侵蝕和膨脹,Pattern Recognition Letters,第 13 卷,第 3 期,1992 年,第 161-166 頁。DOI:10.1016/0167-8655(92)90055-5
- skimage.morphology.isotropic_dilation(image, radius, out=None, spacing=None)[原始碼]#
回傳影像的二元形態學膨脹。
此函式返回與
skimage.morphology.binary_dilation()相同的结果,但在處理大型圓形結構元素時執行速度更快。其原理是對反轉圖像的精確歐幾里得距離圖應用閾值 [1], [2]。該實作基於:func:scipy.ndimage.distance_transform_edt。- 參數:
- imagendarray
二值輸入影像。
- radiusfloat
區域應膨脹的半徑。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- spacingfloat 或 float 序列,可選
沿每個維度元素的間距。如果為序列,則長度必須等於輸入的維度(軸數)。如果為單個數字,則此值會用於所有軸。如果未指定,則表示格點間距為 1。
- 回傳:
- 膨脹後影像布林值的 ndarray
形態學膨脹的結果,值在
[False, True]中。
參考文獻
[1]Cuisenaire, O. 和 Macq, B.,“使用透過傳播的局部距離轉換的快速歐幾里得形態運算子及其應用”,1999 年影像處理及其應用,第七屆國際會議(會議出版物第 465 號),1999 年,第 856-860 頁,第 2 卷。DOI:10.1049/cp:19990446
[2]Ingemar Ragnemalm, 透過輪廓處理和距離圖的閾值處理實現的快速侵蝕和膨脹,Pattern Recognition Letters,第 13 卷,第 3 期,1992 年,第 161-166 頁。DOI:10.1016/0167-8655(92)90055-5
- skimage.morphology.isotropic_erosion(image, radius, out=None, spacing=None)[原始碼]#
回傳影像的二元形態學侵蝕。
此函式返回與
skimage.morphology.binary_erosion()相同的结果,但在處理大型圓形結構元素時執行速度更快。其原理是對圖像的精確歐幾里得距離圖應用閾值 [1], [2]。該實作基於:func:scipy.ndimage.distance_transform_edt。- 參數:
- imagendarray
二值輸入影像。
- radiusfloat
區域應侵蝕的半徑。
- outbool 的 ndarray,可選
用於儲存形態學結果的陣列。如果為 None,則將會分配一個新的陣列。
- spacingfloat 或 float 序列,可選
沿每個維度元素的間距。如果為序列,則長度必須等於輸入的維度(軸數)。如果為單個數字,則此值會用於所有軸。如果未指定,則表示格點間距為 1。
- 回傳:
- 侵蝕後影像布林值的 ndarray
形態學侵蝕的結果,其值在
[False, True]之間。
參考文獻
[1]Cuisenaire, O. 和 Macq, B.,“使用透過傳播的局部距離轉換的快速歐幾里得形態運算子及其應用”,1999 年影像處理及其應用,第七屆國際會議(會議出版物第 465 號),1999 年,第 856-860 頁,第 2 卷。DOI:10.1049/cp:19990446
[2]Ingemar Ragnemalm, 透過輪廓處理和距離圖的閾值處理實現的快速侵蝕和膨脹,Pattern Recognition Letters,第 13 卷,第 3 期,1992 年,第 161-166 頁。DOI:10.1016/0167-8655(92)90055-5
- skimage.morphology.isotropic_opening(image, radius, out=None, spacing=None)[原始碼]#
回傳影像的二元形態學開啟。
此函式返回與
skimage.morphology.binary_opening()相同的结果,但在處理大型圓形結構元素時執行速度更快。其原理是對精確歐幾里得距離圖應用閾值 [1], [2]。該實作基於:func:scipy.ndimage.distance_transform_edt。- 參數:
- imagendarray
二值輸入影像。
- radiusfloat
區域應進行開運算的半徑。
- outbool 的 ndarray,可選
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- spacingfloat 或 float 序列,可選
沿每個維度元素的間距。如果為序列,則長度必須等於輸入的維度(軸數)。如果為單個數字,則此值會用於所有軸。如果未指定,則表示格點間距為 1。
- 回傳:
- 開運算後影像布林值的 ndarray
形態學開運算的結果。
參考文獻
[1]Cuisenaire, O. 和 Macq, B.,“使用透過傳播的局部距離轉換的快速歐幾里得形態運算子及其應用”,1999 年影像處理及其應用,第七屆國際會議(會議出版物第 465 號),1999 年,第 856-860 頁,第 2 卷。DOI:10.1049/cp:19990446
[2]Ingemar Ragnemalm, 透過輪廓處理和距離圖的閾值處理實現的快速侵蝕和膨脹,Pattern Recognition Letters,第 13 卷,第 3 期,1992 年,第 161-166 頁。DOI:10.1016/0167-8655(92)90055-5
- skimage.morphology.label(label_image, background=None, return_num=False, connectivity=None)[原始碼]#
標記整數陣列的連通區域。
當兩個像素是相鄰且具有相同的值時,它們是相連的。在 2D 中,它們可以是 1 連接或 2 連接的相鄰關係。該值是指考慮像素/體素為鄰居的最大正交跳躍次數。
1-connectivity 2-connectivity diagonal connection close-up [ ] [ ] [ ] [ ] [ ] | \ | / | <- hop 2 [ ]--[x]--[ ] [ ]--[x]--[ ] [x]--[ ] | / | \ hop 1 [ ] [ ] [ ] [ ]
- 參數:
- 標籤影像dtype 為 int 的 ndarray
要標記的影像。
- 背景int,可選
將所有具有此值的像素視為背景像素,並將它們標記為 0。預設情況下,值為 0 的像素被視為背景像素。
- return_numbool,可選
是否返回分配的標籤數量。
- connectivity整數,選項
考慮像素/體素為鄰居的最大正交跳躍次數。接受的值範圍從 1 到 input.ndim。如果
None,則使用input.ndim的完整連接性。
- 回傳:
- 標籤dtype 為 int 的 ndarray
已標記的陣列,其中所有相連區域都被分配相同的整數值。
- numint,可選
標籤的數量,等於最大標籤索引,且只有在 return_num 為
True時才會返回。
參考文獻
[1]Christophe Fiorio and Jens Gustedt, “Two linear time Union-Find strategies for image processing”, Theoretical Computer Science 154 (1996), pp. 165-181.
[2]Kensheng Wu, Ekow Otoo and Arie Shoshani, “Optimizing connected component labeling algorithms”, Paper LBNL-56864, 2005, Lawrence Berkeley National Laboratory (University of California), http://repositories.cdlib.org/lbnl/LBNL-56864
範例
>>> import numpy as np >>> x = np.eye(3).astype(int) >>> print(x) [[1 0 0] [0 1 0] [0 0 1]] >>> print(label(x, connectivity=1)) [[1 0 0] [0 2 0] [0 0 3]] >>> print(label(x, connectivity=2)) [[1 0 0] [0 1 0] [0 0 1]] >>> print(label(x, background=-1)) [[1 2 2] [2 1 2] [2 2 1]] >>> x = np.array([[1, 0, 0], ... [1, 1, 5], ... [0, 0, 0]]) >>> print(label(x)) [[1 0 0] [1 1 2] [0 0 0]]
- skimage.morphology.local_maxima(image, footprint=None, connectivity=None, indices=False, allow_borders=True)[原始碼]#
尋找 n 維陣列的局部最大值。
局部最大值被定義為具有相等灰度級(平坦區)的相連像素集合,其灰度級嚴格大於鄰域中所有像素的灰度級。
- 參數:
- imagendarray
一個 n 維陣列。
- footprintndarray,選項
用於確定每個評估像素鄰域的足跡(結構元素)(
True表示相連的像素)。它必須是一個布林陣列,且維度數必須與image相同。如果沒有給定footprint或connectivity,則所有相鄰像素都會被視為鄰域的一部分。- connectivity整數,選項
一個用於確定每個評估像素的鄰域的數字。與中心平方距離小於或等於
connectivity的相鄰像素被視為鄰居。如果footprint不是 None,則會忽略。- indicesbool,可選
如果為 True,則輸出將是一個一維陣列的元組,表示每個維度中局部最大值的索引。如果為 False,則輸出將是一個與
image具有相同形狀的布林陣列。- allow_bordersbool,可選
如果為 true,則接觸影像邊界的平坦區為有效最大值。
- 回傳:
- maximandarray 或 tuple[ndarray]
如果
indices為 false,則會返回一個與image具有相同形狀的布林陣列,其中True表示局部最大值的位置(否則為False)。如果indices為 true,則會返回一個一維陣列的元組,其中包含所有找到的最大值的座標(索引)。
- 警告:
- UserWarning
如果
allow_borders為 false,且給定image的任何維度短於 3 個樣本,則最大值不存在,並會顯示警告。
注意
此函式基於以下概念運作
對影像的最後一個維度進行第一次掃描,並僅通過比較一個方向上的像素來標記局部最大值的候選像素。如果像素在最後一個維度中沒有相連,則所有像素都會被標記為候選像素。
針對每個候選像素
執行洪水填充,以找到所有具有相同灰度值且屬於平坦區的相連像素。
考慮平坦區的相連鄰域:如果沒有邊界的樣本具有更高的灰度級,則將該平坦區標記為確定的局部最大值。
範例
>>> from skimage.morphology import local_maxima >>> image = np.zeros((4, 7), dtype=int) >>> image[1:3, 1:3] = 1 >>> image[3, 0] = 1 >>> image[1:3, 4:6] = 2 >>> image[3, 6] = 3 >>> image array([[0, 0, 0, 0, 0, 0, 0], [0, 1, 1, 0, 2, 2, 0], [0, 1, 1, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
通過與所有相鄰像素比較來查找局部最大值(最大連接性)
>>> local_maxima(image) array([[False, False, False, False, False, False, False], [False, True, True, False, False, False, False], [False, True, True, False, False, False, False], [ True, False, False, False, False, False, True]]) >>> local_maxima(image, indices=True) (array([1, 1, 2, 2, 3, 3]), array([1, 2, 1, 2, 0, 6]))
在不與對角像素比較的情況下查找局部最大值(連接性 1)
>>> local_maxima(image, connectivity=1) array([[False, False, False, False, False, False, False], [False, True, True, False, True, True, False], [False, True, True, False, True, True, False], [ True, False, False, False, False, False, True]])
並排除與影像邊緣接壤的最大值
>>> local_maxima(image, connectivity=1, allow_borders=False) array([[False, False, False, False, False, False, False], [False, True, True, False, True, True, False], [False, True, True, False, True, True, False], [False, False, False, False, False, False, False]])
- skimage.morphology.local_minima(image, footprint=None, connectivity=None, indices=False, allow_borders=True)[原始碼]#
尋找 n 維陣列的局部最小值。
局部最小值定義為具有相同灰度級(平坦區)的相連像素集合,且該灰度級嚴格小於鄰域中所有像素的灰度級。
- 參數:
- imagendarray
一個 n 維陣列。
- footprintndarray,選項
用於確定每個評估像素鄰域的足跡(結構元素)(
True表示相連的像素)。它必須是一個布林陣列,且維度數必須與image相同。如果沒有給定footprint或connectivity,則所有相鄰像素都會被視為鄰域的一部分。- connectivity整數,選項
一個用於確定每個評估像素的鄰域的數字。與中心平方距離小於或等於
connectivity的相鄰像素被視為鄰居。如果footprint不是 None,則會忽略。- indicesbool,可選
若為 True,則輸出將是一個一維陣列的元組,表示每個維度中局部最小值的索引。若為 False,則輸出將是一個布林陣列,其形狀與
image相同。- allow_bordersbool,可選
若為 true,則接觸影像邊界的平坦區視為有效的最小值。
- 回傳:
- minimandarray 或 tuple[ndarray]
若
indices為 false,則會返回一個布林陣列,其形狀與image相同,其中True表示局部最小值的位置(否則為False)。若indices為 true,則會返回一個一維陣列的元組,其中包含所有找到的最小值的座標(索引)。
注意
此函式基於以下概念運作
對影像的最後一個維度進行第一次掃描,並僅比較一個方向上的像素,以標記局部最小值的候選者。如果像素在最後一個維度中沒有連接,則會將所有像素都標記為候選者。
針對每個候選像素
執行洪水填充,以找到所有具有相同灰度值且屬於平坦區的相連像素。
考慮平坦區的相連鄰域:如果沒有邊界樣本的灰度級較小,則將平坦區標記為確定的局部最小值。
範例
>>> from skimage.morphology import local_minima >>> image = np.zeros((4, 7), dtype=int) >>> image[1:3, 1:3] = -1 >>> image[3, 0] = -1 >>> image[1:3, 4:6] = -2 >>> image[3, 6] = -3 >>> image array([[ 0, 0, 0, 0, 0, 0, 0], [ 0, -1, -1, 0, -2, -2, 0], [ 0, -1, -1, 0, -2, -2, 0], [-1, 0, 0, 0, 0, 0, -3]])
透過與所有相鄰像素(最大連通性)比較來尋找局部最小值
>>> local_minima(image) array([[False, False, False, False, False, False, False], [False, True, True, False, False, False, False], [False, True, True, False, False, False, False], [ True, False, False, False, False, False, True]]) >>> local_minima(image, indices=True) (array([1, 1, 2, 2, 3, 3]), array([1, 2, 1, 2, 0, 6]))
尋找局部最小值而不與對角像素(連通性 1)比較
>>> local_minima(image, connectivity=1) array([[False, False, False, False, False, False, False], [False, True, True, False, True, True, False], [False, True, True, False, True, True, False], [ True, False, False, False, False, False, True]])
並排除與影像邊緣相鄰的最小值
>>> local_minima(image, connectivity=1, allow_borders=False) array([[False, False, False, False, False, False, False], [False, True, True, False, True, True, False], [False, True, True, False, True, True, False], [False, False, False, False, False, False, False]])
- skimage.morphology.max_tree(image, connectivity=1)[原始碼]#
從影像建立最大樹。
元件樹表示透過應用於影像的連續閾值操作所產生的相連元件的階層結構。如果較高層級的元件包含在較低層級的元件中,則較低層級的相連元件是較高層級元件的父元件。最大樹是元件樹的有效表示。較低層級的相連元件由該層級的一個參考像素表示,該參考像素是該層級所有其他像素以及較高層級參考像素的父像素。最大樹是許多形態學運算子(即相連運算子)的基礎。
- 參數:
- imagendarray
要計算最大樹的輸入影像。此影像可以是任何類型。
- connectivity無號整數,選用
鄰域連通性。整數表示到達鄰居的最大正交步數。在 2D 中,4 鄰域為 1,而 8 鄰域為 2。預設值為 1。
- 回傳:
- parentndarray,int64
形狀與影像相同的陣列。每個像素的值是其在攤平陣列中的父項索引。
- tree_traverser一維陣列,int64
已排序的像素索引(參考展開的陣列)。像素的排序方式為每個像素都位於其父項之前(根除外,根沒有父項)。
參考文獻
[1]Salembier, P., Oliveras, A., & Garrido, L. (1998)。用於影像和序列處理的非擴張連通運算子。IEEE 影像處理交易,7(4),555-570。DOI:10.1109/83.663500
[2]Berger, C., Geraud, T., Levillain, R., Widynski, N., Baillard, A., Bertin, E. (2007). Effective Component Tree Computation with Application to Pattern Recognition in Astronomical Imaging. In International Conference on Image Processing (ICIP) (pp. 41-44). DOI:10.1109/ICIP.2007.4379949
[3]Najman, L., & Couprie, M. (2006)。以擬線性時間建立元件樹。IEEE 影像處理交易,15(11),3531-3539。DOI:10.1109/TIP.2006.877518
[4]Carlinet, E., & Geraud, T. (2014)。元件樹計算演算法的比較評論。IEEE 影像處理交易,23(9),3885-3895。DOI:10.1109/TIP.2014.2336551
範例
我們建立一個小的範例影像(來自 [4] 的圖 1),並建立最大樹。
>>> image = np.array([[15, 13, 16], [12, 12, 10], [16, 12, 14]]) >>> P, S = max_tree(image, connectivity=2)
- skimage.morphology.max_tree_local_maxima(image, connectivity=1, parent=None, tree_traverser=None)[原始碼]#
決定影像的所有局部最大值。
局部最大值定義為具有相同灰度級的相連像素集合,且該灰度級嚴格大於該集合的直接鄰域中所有像素的灰度級。該函式會標記局部最大值。
從技術上講,此實作基於影像的最大樹表示。如果已計算最大樹表示,則此函式非常有效率。否則,最好使用函式 local_maxima。
- 參數:
- imagendarray
要計算最大值的輸入影像。
- connectivity無號整數,選用
鄰域連通性。整數表示到達鄰居的最大正交步數。在 2D 中,4 鄰域為 1,而 8 鄰域為 2。預設值為 1。
- parentndarray, int64,選用
每個像素的值是其在攤平陣列中的父項索引。
- tree_traverser1D 陣列,int64,選用
已排序的像素索引(參考展開的陣列)。像素的排序方式為每個像素都位於其父項之前(根除外,根沒有父項)。
- 回傳:
- local_maxndarray,uint64
影像的已標記局部最大值。
參考文獻
[1]Vincent L., Proc. “灰階面積開啟和閉合,其有效實作和應用”,EURASIP 數學形態學及其在訊號處理的應用研討會,西班牙巴塞隆納,第 22-27 頁,1993 年 5 月。
[2]Soille, P., “形態學影像分析:原理和應用”(第 6 章),第 2 版 (2003),ISBN 3540429883。DOI:10.1007/978-3-662-05088-0
[3]Salembier, P., Oliveras, A., & Garrido, L. (1998)。用於影像和序列處理的非擴張連通運算子。IEEE 影像處理交易,7(4),555-570。DOI:10.1109/83.663500
[4]Najman, L., & Couprie, M. (2006)。以擬線性時間建立元件樹。IEEE 影像處理交易,15(11),3531-3539。DOI:10.1109/TIP.2006.877518
[5]Carlinet, E., & Geraud, T. (2014)。元件樹計算演算法的比較評論。IEEE 影像處理交易,23(9),3885-3895。DOI:10.1109/TIP.2014.2336551
範例
我們建立一個影像(中心具有最大值的二次函式,以及 4 個額外的恆定最大值)。
>>> w = 10 >>> x, y = np.mgrid[0:w,0:w] >>> f = 20 - 0.2*((x - w/2)**2 + (y-w/2)**2) >>> f[2:4,2:4] = 40; f[2:4,7:9] = 60; f[7:9,2:4] = 80; f[7:9,7:9] = 100 >>> f = f.astype(int)
我們可以計算所有局部最大值
>>> maxima = max_tree_local_maxima(f)
產生的影像包含已標記的局部最大值。
- skimage.morphology.medial_axis(image, mask=None, return_distance=False, *, rng=None)[原始碼]#
計算二元影像的中軸轉換。
- 參數:
- image二元 ndarray,形狀 (M, N)
要骨架化的形狀的影像。如果此輸入還不是二元影像,則會將其轉換為二元影像:在這種情況下,零值被視為背景 (False),非零值被視為前景 (True)。
- mask二元 ndarray,形狀 (M, N),選用
如果給定遮罩,則僅使用
image中在mask中具有 true 值的那些元素來計算中軸。- return_distance布林值,選用
如果為 true,則會返回距離轉換以及骨架。
- rng{
numpy.random.Generator,int},選用 虛擬隨機數產生器。預設情況下,會使用 PCG64 產生器(請參閱
numpy.random.default_rng())。如果rng是一個 int,則會使用它來初始化產生器。PRNG 決定了處理像素以打破平手的順序。
在 0.19 版中新增。
- 回傳:
- out布林值的 ndarray
影像的中軸轉換
- dist整數的 ndarray,選用
影像的距離轉換(僅在
return_distance為 True 時返回)
另請參閱
注意
此演算法將影像的中軸轉換計算為其距離轉換的脊線。
- 該演算法的不同步驟如下
使用查閱表,該表會將 0 或 1 指派給 3x3 二元方格的每個配置,無論是否應該移除或保留中心像素。如果某個點有多個鄰居,且移除該點不會變更相連元件的數量,則我們希望移除該點。
計算到背景的距離轉換,以及像素的角點性。
前景(值為 1)點按照距離轉換,然後按照角點性排序。
呼叫一個 cython 函式將影像縮減為骨架。它會按照上一步確定的順序處理像素,並根據查閱表移除或保留像素。由於排序,因此可以在一次掃描中處理所有像素。
範例
>>> square = np.zeros((7, 7), dtype=bool) >>> square[1:-1, 2:-2] = 1 >>> square.view(np.uint8) array([[0, 0, 0, 0, 0, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 0, 0, 0, 0, 0]], dtype=uint8) >>> medial_axis(square).view(np.uint8) array([[0, 0, 0, 0, 0, 0, 0], [0, 0, 1, 0, 1, 0, 0], [0, 0, 0, 1, 0, 0, 0], [0, 0, 0, 1, 0, 0, 0], [0, 0, 0, 1, 0, 0, 0], [0, 0, 1, 0, 1, 0, 0], [0, 0, 0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.mirror_footprint(footprint)[原始碼]#
鏡像足跡中的每個維度。
- 參數:
- footprintndarray 或 tuple
輸入足跡或足跡序列
- 回傳:
- invertedndarray 或 tuple
沿每個維度鏡像的足跡。
範例
>>> footprint = np.array([[0, 0, 0], ... [0, 1, 1], ... [0, 1, 1]], np.uint8) >>> mirror_footprint(footprint) array([[1, 1, 0], [1, 1, 0], [0, 0, 0]], dtype=uint8)
- skimage.morphology.octagon(m, n, dtype=<class 'numpy.uint8'>, *, decomposition=None)[原始碼]#
產生八邊形足跡。
對於給定的 (m) 水平和垂直邊長,以及給定的 (n) 斜邊高度或寬度,將會產生一個八邊形。斜邊與水平軸的夾角為 45 度或 135 度,因此寬度和高度相等。沿單一軸的整體佔用空間大小將會是
m + 2 * n。- 參數:
- mint
水平和垂直邊的長度。
- nint
斜邊的高度或寬度。
- 回傳:
- footprintndarray 或 tuple
足跡,其中鄰域的元素為 1,否則為 0。當
decomposition為 None 時,這只是一個 numpy.ndarray。否則,這會是一個 tuple,其長度等於要套用的唯一結構元素的數量(請參閱「注意事項」以取得更多詳細資訊)
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- decomposition{None, 'sequence'},可選
如果為 None,則會傳回單一陣列。對於 ‘sequence’,會傳回較小足跡的 tuple。套用這一系列的較小足跡,會得到與單一較大足跡相同的結果,但具有更好的計算效能。請參閱「注意事項」以取得更多詳細資訊。
注意
當
decomposition不為 None 時,footprinttuple 的每個元素都是(ndarray, num_iter)形式的 2-tuple,指定足跡陣列及其要套用的疊代次數。對於二元或灰階形態學,觀察到使用
decomposition='sequence'具有效能優勢,且優勢的大小隨著足跡大小的增加而增加。
- skimage.morphology.octahedron(radius, dtype=<class 'numpy.uint8'>, *, decomposition=None)[原始碼]#
產生八面體足跡。
這是菱形的 3D 等效形式。如果像素與鄰域中心之間的城市街區/曼哈頓距離不大於半徑,則該像素是鄰域的一部分(即標記為 1)。
- 參數:
- radius整數
八面體形狀的佔用空間半徑。
- 回傳:
- footprintndarray 或 tuple
足跡,其中鄰域的元素為 1,否則為 0。當
decomposition為 None 時,這只是一個 numpy.ndarray。否則,這會是一個 tuple,其長度等於要套用的唯一結構元素的數量(請參閱「注意事項」以取得更多詳細資訊)
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- decomposition{None, 'sequence'},可選
如果為 None,則會傳回單一陣列。對於 ‘sequence’,會傳回較小足跡的 tuple。套用這一系列的較小足跡,會得到與單一較大足跡相同的結果,但具有更好的計算效能。請參閱「注意事項」以取得更多詳細資訊。
注意
當
decomposition不為 None 時,footprinttuple 的每個元素都是(ndarray, num_iter)形式的 2-tuple,指定足跡陣列及其要套用的疊代次數。對於二元或灰階形態學,觀察到使用
decomposition='sequence'具有效能優勢,且優勢的大小隨著足跡大小的增加而增加。
- skimage.morphology.opening(image, footprint=None, out=None, *, mode='reflect', cval=0.0)[原始碼]#
回傳影像的灰階形態學開啟。
影像的形態學開運算定義為先腐蝕後膨脹。開運算可以移除小的亮點(即「鹽」)並連接小的暗裂縫。這往往會「打開」(暗)特徵之間的間隙。
- 參數:
- imagendarray
影像陣列。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outndarray,選用
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效的模式為:‘reflect’、‘constant’、‘nearest’、‘mirror’、‘wrap’、‘max’、‘min’ 或 ‘ignore’。如果為 ‘ignore’,則假設影像範圍外的像素在侵蝕中為影像 dtype 的最大值,而在膨脹中則為最小值,這會導致它們不影響結果。預設值為 ‘reflect’。- cval純量,選用
如果
mode為 ‘constant’,則填充輸入邊緣外的值。預設值為 0.0。在 0.23 版中新增:
mode和cval是在 0.23 版中新增的。
- 回傳:
- opening陣列,與
image具有相同的形狀和類型 形態學開運算的結果。
- opening陣列,與
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。範例
>>> # Open up gap between two bright regions (but also shrink regions) >>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> bad_connection = np.array([[1, 0, 0, 0, 1], ... [1, 1, 0, 1, 1], ... [1, 1, 1, 1, 1], ... [1, 1, 0, 1, 1], ... [1, 0, 0, 0, 1]], dtype=np.uint8) >>> opening(bad_connection, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [1, 1, 0, 1, 1], [1, 1, 0, 1, 1], [1, 1, 0, 1, 1], [0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.pad_footprint(footprint, *, pad_end=True)[原始碼]#
沿著每個維度將足跡填充為奇數大小。
- 參數:
- footprintndarray 或 tuple
輸入足跡或足跡序列
- pad_endbool,可選
如果
True,則在每個維度的末尾(右側)填充,否則在前面(左側)填充。
- 回傳:
- paddedndarray 或 tuple
佔用空間,沿每個維度填充為奇數大小。
範例
>>> footprint = np.array([[0, 0], ... [1, 1], ... [1, 1]], np.uint8) >>> pad_footprint(footprint) array([[0, 0, 0], [1, 1, 0], [1, 1, 0]], dtype=uint8)
- skimage.morphology.reconstruction(seed, mask, method='dilation', footprint=None, offset=None)[原始碼]#
執行影像的形態學重建。
通過膨脹進行形態學重建類似於基本的形態學膨脹:高強度值將取代附近低強度值。然而,基本的膨脹運算子會使用佔用空間來決定輸入影像中的值可以擴散多遠。相反地,重建使用兩個影像:「種子」影像,指定擴散的值;以及「遮罩」影像,給出每個像素允許的最大值。遮罩影像就像佔用空間一樣,限制了高強度值的擴散。通過腐蝕進行重建只是簡單的反向操作:低強度值從種子影像擴散,並受到遮罩影像的限制,遮罩影像表示允許的最小值。
或者,您可以將重建視為隔離影像連接區域的一種方式。對於膨脹,重建會連接種子影像中局部最大值標記的區域:與這些種子小於或等於的相鄰像素會連接到種子區域。值大於種子影像的局部最大值將被截斷為種子值。
- 參數:
- seedndarray
種子影像(又稱標記影像),指定膨脹或腐蝕的值。
- maskndarray
每個像素允許的最大值(膨脹)/最小值(腐蝕)。
- method{‘dilation’|’erosion’},可選
通過膨脹或腐蝕執行重建。在膨脹(或腐蝕)中,種子影像會膨脹(或腐蝕),直到受到遮罩影像的限制。對於膨脹,每個種子值必須小於或等於相應的遮罩值;對於腐蝕,情況則相反。預設值為「膨脹」。
- footprintndarray,選項
鄰域表示為 1 和 0 的 n 維陣列。預設值為半徑等於 1 的 n 維正方形(即 2D 影像為 3x3 正方形,3D 影像為 3x3x3 立方體等)。
- offsetndarray,可選
佔用空間中心的座標。預設值位於佔用空間的幾何中心,在這種情況下,佔用空間的維度必須是奇數。
- 回傳:
- reconstructedndarray
形態學重建的結果。
注意
該演算法取自 [1]。關於灰度重建的應用在 [2] 和 [3] 中進行了討論。
參考文獻
[1]Robinson,“高效形態學重建:下坡過濾器”,《模式識別快報》25 (2004) 1759-1767。
[2]Vincent, L.,“影像分析中的形態學灰度重建:應用和高效演算法”,《IEEE 影像處理學報》(1993)
[3]Soille, P.,“形態學影像分析:原理和應用”,第 6 章,第 2 版 (2003),ISBN 3540429883。
範例
>>> import numpy as np >>> from skimage.morphology import reconstruction
首先,我們建立一個正弦遮罩影像,其峰值位於中間和末端。
>>> x = np.linspace(0, 4 * np.pi) >>> y_mask = np.cos(x)
然後,我們建立一個初始化為最小遮罩值的種子影像(對於通過膨脹進行重建,最小強度值不會擴散),並在左側和右側峰值添加「種子」,但其值為峰值 (1) 的一小部分。
>>> y_seed = y_mask.min() * np.ones_like(x) >>> y_seed[0] = 0.5 >>> y_seed[-1] = 0 >>> y_rec = reconstruction(y_seed, y_mask)
重建的影像(或本例中的曲線)與遮罩影像完全相同,只是峰值被截斷為 0.5 和 0。中間峰值完全消失:由於此峰值區域中沒有種子值,因此其重建值被截斷為周圍的值 (-1)。
作為一個更實際的範例,我們嘗試通過減去由重建建立的背景影像來提取影像的明亮特徵。
>>> y, x = np.mgrid[:20:0.5, :20:0.5] >>> bumps = np.sin(x) + np.sin(y)
為了建立背景影像,將遮罩影像設定為原始影像,並將種子影像設定為具有強度偏移
h的原始影像。>>> h = 0.3 >>> seed = bumps - h >>> background = reconstruction(seed, bumps)
產生的重建影像看起來與原始影像完全相同,但凸起的峰值被截斷。從原始影像中減去此重建影像只會留下凸起的峰值
>>> hdome = bumps - background
此運算稱為影像的 h-dome,並在減去的影像中留下高度
h的特徵。
- skimage.morphology.remove_objects_by_distance(label_image, min_distance, *, priority=None, p_norm=2, spacing=None, out=None)[原始碼]#
按照指定的順序移除物件,直到剩餘物件保持最小距離。
從影像中移除標記的物件,直到剩餘的物件彼此之間的間距大於給定的距離。預設情況下,會先移除較小的物件。
- 參數:
- label_image整數的 ndarray
一個包含物件標籤的 n 維陣列,例如由
label()傳回的陣列。值為零被視為背景,所有其他物件 ID 必須為正整數。- min_distanceint 或 float
移除與其他物件的距離不超過此正值的物件。會先移除
priority較低的物件。- priorityndarray,可選
定義移除物件的優先順序。需要一個長度為
np.amax(label_image) + 1的一維陣列,該陣列包含每個物件標籤在其各自索引處的優先順序。會先移除值較低的物件,直到所有剩餘物件都滿足距離要求。如果未給定,則優先順序會給予樣本數量較多的物件,其次是其標籤值。- p_normint 或 float,可選
用於計算物件之間距離的 p 階 Minkowski 距離。預設值
2對應於歐幾里得距離,1對應於「曼哈頓」距離,np.inf對應於切比雪夫距離。- spacing浮點數序列,可選
label_image的每個軸上的像素間距。如果未指定,則表示網格間距為 1。- outndarray,選用
與
image具有相同形狀和 dtype 的陣列,輸出結果將放置於此。預設情況下,將會建立一個新的陣列。
- 回傳:
- outndarray
與
label_image具有相同形狀的陣列,其中違反min_distance條件的物件已被移除。
另請參閱
skimage.morphology.remove_small_objects移除小於指定大小的物件。
注意
此演算法的基本步驟如下:
找出所有給定物件的索引,並根據它們是否指向物件的邊界進行分離。
依據標籤值對索引進行排序,確保指向同一物件的索引彼此相鄰。此最佳化可讓您只需逐步移動到相鄰的索引,即可找到物件的所有部分。
依據
priority對邊界索引進行排序。使用穩定排序,以保留先前排序步驟的順序。如果未給定priority,則將使用numpy.bincount()作為後備。從邊界索引建構
scipy.spatial.cKDTree。依據優先排序的順序,迭代遍歷邊界索引,並查詢 kd 樹以找出太接近的物件。移除這些物件,並且在稍後評估其他物件時不將它們納入考量。
此演算法的效能取決於
label_image中屬於物件邊界的樣本數量。範例
>>> import skimage as ski >>> ski.morphology.remove_objects_by_distance(np.array([2, 0, 1, 1]), 2) array([0, 0, 1, 1]) >>> ski.morphology.remove_objects_by_distance( ... np.array([2, 0, 1, 1]), 2, priority=np.array([0, 1, 9]) ... ) array([2, 0, 0, 0]) >>> label_image = np.array( ... [[8, 0, 0, 0, 0, 0, 0, 0, 0, 9, 9], ... [8, 8, 8, 0, 0, 0, 0, 0, 0, 9, 9], ... [0, 0, 0, 0, 0, 0, 0, 0, 9, 0, 0], ... [0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0], ... [0, 0, 3, 0, 0, 0, 1, 0, 0, 0, 0], ... [2, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0, 0, 0, 0, 0, 7, 7]] ... ) >>> ski.morphology.remove_objects_by_distance( ... label_image, min_distance=3 ... ) array([[8, 0, 0, 0, 0, 0, 0, 0, 0, 9, 9], [8, 8, 8, 0, 0, 0, 0, 0, 0, 9, 9], [0, 0, 0, 0, 0, 0, 0, 0, 9, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [2, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 7, 7]])
- skimage.morphology.remove_small_holes(ar, area_threshold=64, connectivity=1, *, out=None)[來源]#
移除小於指定大小的連續孔洞。
- 參數:
- arndarray(任意形狀,int 或 bool 型別)
包含感興趣的連通元件的陣列。
- area_thresholdint,可選(預設值:64)
將被填滿的連續孔洞的最大面積(以像素為單位)。取代
min_size。- connectivityint,{1, 2, …, ar.ndim},可選(預設值:1)
定義像素鄰域的連通性。
- outndarray
與
ar具有相同形狀和 bool dtype 的陣列,輸出結果將放置於此。預設情況下,將會建立一個新的陣列。
- 回傳:
- outndarray,與輸入
ar具有相同的形狀和類型 已移除連通元件內小孔的輸入陣列。
- outndarray,與輸入
- 引發:
- TypeError
如果輸入陣列為無效類型,例如浮點數或字串。
- ValueError
如果輸入陣列包含負值。
注意
如果陣列類型為 int,則假設其已包含已標記的物件。標籤不會保留在輸出影像中(此函數始終輸出 bool 影像)。建議在使用此函數後完成標記。
範例
>>> from skimage import morphology >>> a = np.array([[1, 1, 1, 1, 1, 0], ... [1, 1, 1, 0, 1, 0], ... [1, 0, 0, 1, 1, 0], ... [1, 1, 1, 1, 1, 0]], bool) >>> b = morphology.remove_small_holes(a, 2) >>> b array([[ True, True, True, True, True, False], [ True, True, True, True, True, False], [ True, False, False, True, True, False], [ True, True, True, True, True, False]]) >>> c = morphology.remove_small_holes(a, 2, connectivity=2) >>> c array([[ True, True, True, True, True, False], [ True, True, True, False, True, False], [ True, False, False, True, True, False], [ True, True, True, True, True, False]]) >>> d = morphology.remove_small_holes(a, 2, out=a) >>> d is a True
- skimage.morphology.remove_small_objects(ar, min_size=64, connectivity=1, *, out=None)[來源]#
移除小於指定大小的物件。
預期 ar 為具有已標記物件的陣列,並移除小於 min_size 的物件。如果
ar為 bool,則會先標記影像。這會導致 bool 和 0 與 1 陣列可能出現不同的行為。- 參數:
- arndarray(任意形狀,int 或 bool 型別)
包含感興趣物件的陣列。如果陣列類型為 int,則整數必須為非負數。
- min_sizeint,可選(預設值:64)
最小可允許的物件大小。
- connectivityint,{1, 2, …, ar.ndim},可選(預設值:1)
定義像素鄰域的連通性。如果
ar為 bool,則在標記期間使用。- outndarray
與
ar具有相同形狀的陣列,輸出結果將放置於此。預設情況下,將會建立一個新的陣列。
- 回傳:
- outndarray,與輸入
ar具有相同的形狀和類型 已移除小連通元件的輸入陣列。
- outndarray,與輸入
- 引發:
- TypeError
如果輸入陣列為無效類型,例如浮點數或字串。
- ValueError
如果輸入陣列包含負值。
範例
>>> from skimage import morphology >>> a = np.array([[0, 0, 0, 1, 0], ... [1, 1, 1, 0, 0], ... [1, 1, 1, 0, 1]], bool) >>> b = morphology.remove_small_objects(a, 6) >>> b array([[False, False, False, False, False], [ True, True, True, False, False], [ True, True, True, False, False]]) >>> c = morphology.remove_small_objects(a, 7, connectivity=2) >>> c array([[False, False, False, True, False], [ True, True, True, False, False], [ True, True, True, False, False]]) >>> d = morphology.remove_small_objects(a, 6, out=a) >>> d is a True
- skimage.morphology.skeletonize(image, *, method=None)[來源]#
透過細化計算輸入影像的骨骼。
- 參數:
- 回傳:
- skeleton(M, N[, P]) ndarray,bool 型別
細化後的影像。
另請參閱
參考文獻
[Lee94]T.-C. Lee、R.L. Kashyap 和 C.-N. Chu,「Building skeleton models via 3-D medial surface/axis thinning algorithms」,《Computer Vision, Graphics, and Image Processing》,56(6):462-478, 1994。
[Zha84]T. Y. Zhang 和 C. Y. Suen,「A fast parallel algorithm for thinning digital patterns」,《Communications of the ACM》,1984 年 3 月,第 27 卷,第 3 期。
範例
>>> X, Y = np.ogrid[0:9, 0:9] >>> ellipse = (1./3 * (X - 4)**2 + (Y - 4)**2 < 3**2).astype(bool) >>> ellipse.view(np.uint8) array([[0, 0, 0, 1, 1, 1, 0, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 0, 0], [0, 0, 0, 1, 1, 1, 0, 0, 0]], dtype=uint8) >>> skel = skeletonize(ellipse) >>> skel.view(np.uint8) array([[0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 1, 0, 0, 0, 0], [0, 0, 0, 0, 1, 0, 0, 0, 0], [0, 0, 0, 0, 1, 0, 0, 0, 0], [0, 0, 0, 0, 1, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.star(a, dtype=<class 'numpy.uint8'>)[來源]#
產生星形足跡。
星形具有 8 個頂點,是大小為
2*a + 1的正方形與其旋轉 45 度的版本的重疊。傾斜邊與水平軸呈 45 或 135 度。- 參數:
- aint
決定星形結構元素大小的參數。傳回的正方形陣列邊長為
2*a + 1 + 2*floor(a / 2)。
- 回傳:
- footprintndarray
足跡,其中鄰域的元素為 1,否則為 0。
- 其他參數:
- dtype資料類型,可選
足跡的資料類型。
- skimage.morphology.thin(image, max_num_iter=None)[來源]#
執行二元影像的形態學細化。
- 參數:
- image二值 (M, N) ndarray
要細化的影像。如果此輸入還不是二值影像,則會將其轉換為二值影像:在這種情況下,零值被視為背景 (False),非零值被視為前景 (True)。
- max_num_iterint,迭代次數,可選
無論此參數的值為何,如果迭代沒有產生任何變更,則會立即傳回細化後的影像。因此,如果指定此參數,則會設定執行迭代次數的上限。
- 回傳:
- outndarray,bool 型別
細化後的影像。
另請參閱
注意
此演算法 [1] 的運作方式是多次掃過影像,移除符合設計用於細化連通區域的條件集,同時保留八連通元件和 2 x 2 正方形的像素 [2]。在兩個子迭代的每一個中,演算法會將中間骨架影像與鄰域遮罩相關聯,然後在查閱表中查閱每個鄰域,以指出是否應在該子迭代中刪除中心像素。
參考文獻
[1]Z. Guo 和 R. W. Hall,「Parallel thinning with two-subiteration algorithms」,《Comm. ACM》,第 32 卷,第 3 期,第 359-373 頁,1989 年。DOI:10.1145/62065.62074
[2]Lam, L.、Seong-Whan Lee 和 Ching Y. Suen,「Thinning Methodologies-A Comprehensive Survey」,《IEEE Transactions on Pattern Analysis and Machine Intelligence》,第 14 卷,第 9 期,第 879 頁,1992 年。DOI:10.1109/34.161346
範例
>>> square = np.zeros((7, 7), dtype=bool) >>> square[1:-1, 2:-2] = 1 >>> square[0, 1] = 1 >>> square.view(np.uint8) array([[0, 1, 0, 0, 0, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 0, 0], [0, 0, 0, 0, 0, 0, 0]], dtype=uint8) >>> skel = thin(square) >>> skel.view(np.uint8) array([[0, 1, 0, 0, 0, 0, 0], [0, 0, 1, 0, 0, 0, 0], [0, 0, 0, 1, 0, 0, 0], [0, 0, 0, 1, 0, 0, 0], [0, 0, 0, 1, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0]], dtype=uint8)
- skimage.morphology.white_tophat(image, footprint=None, out=None, *, mode='reflect', cval=0.0)[原始碼]#
回傳影像的白色禮帽。
影像的白色禮帽 (white top hat) 定義為影像減去其形態學開運算 (morphological opening) 的結果。此運算會返回影像中比足跡 (footprint) 小的亮點。
- 參數:
- imagendarray
影像陣列。
- footprintndarray 或 tuple,可選
鄰域表示為 1 和 0 的二維陣列。如果為 None,則使用十字形足跡 (connectivity=1)。也可以提供足跡作為較小足跡的序列,如下面注意事項中所述。
- outndarray,選用
儲存形態學結果的陣列。如果傳遞 None,則將分配一個新陣列。
- modestr,可選
mode參數決定如何處理陣列邊界。有效的模式為:'reflect'、'constant'、'nearest'、'mirror'、'wrap'、'max'、'min' 或 'ignore'。請參閱skimage.morphology.opening()。預設值為 'reflect'。- cval純量,選用
如果
mode為 ‘constant’,則填充輸入邊緣外的值。預設值為 0.0。在 0.23 版中新增:
mode和cval是在 0.23 版中新增的。
- 回傳:
- out陣列,與
image具有相同的形狀和類型 形態學白色禮帽運算的結果。
- out陣列,與
另請參閱
注意
足跡也可以作為 2 元組的序列提供,其中每個 2 元組的第一個元素是足跡 ndarray,第二個元素是描述其應該迭代次數的整數。例如,
footprint=[(np.ones((9, 1)), 1), (np.ones((1, 9)), 1)]會先應用 9x1 足跡,然後應用 1x9 足跡,產生的最終效果與footprint=np.ones((9, 9))相同,但計算成本較低。大多數內建足跡(例如skimage.morphology.disk())提供自動產生此類型足跡序列的選項。參考文獻
範例
>>> # Subtract gray background from bright peak >>> import numpy as np >>> from skimage.morphology import footprint_rectangle >>> bright_on_gray = np.array([[2, 3, 3, 3, 2], ... [3, 4, 5, 4, 3], ... [3, 5, 9, 5, 3], ... [3, 4, 5, 4, 3], ... [2, 3, 3, 3, 2]], dtype=np.uint8) >>> white_tophat(bright_on_gray, footprint_rectangle((3, 3))) array([[0, 0, 0, 0, 0], [0, 0, 1, 0, 0], [0, 1, 5, 1, 0], [0, 0, 1, 0, 0], [0, 0, 0, 0, 0]], dtype=uint8)