注意
前往結尾以下載完整的範例程式碼。或通過 Binder 在您的瀏覽器中執行此範例
使用修復技術還原帶有斑點的角膜影像#
光學同調斷層掃描 (OCT) 是一種由眼科醫生使用,用於拍攝患者眼後部的非侵入性影像技術 [1]。在執行 OCT 時,灰塵可能會黏在設備的參考鏡上,導致影像上出現黑點。問題是這些污垢點覆蓋了體內組織的區域,因此隱藏了感興趣的資料。我們這裡的目標是根據邊界附近的像素來還原(重建)隱藏區域。
本教學改編自 Jules Scholler 分享的應用程式 [2]。這些影像是由 Viacheslav Mazlin 獲取的(請參閱 skimage.data.palisades_of_vogt())。
import matplotlib.pyplot as plt
import numpy as np
import plotly.io
import plotly.express as px
import skimage as ski
我們在這裡使用的資料集是人類體內組織的影像序列(一部影片!)。具體而言,它顯示了特定角膜樣本的沃格特柵欄。
載入影像資料#
image_seq = ski.data.palisades_of_vogt()
print(f'number of dimensions: {image_seq.ndim}')
print(f'shape: {image_seq.shape}')
print(f'dtype: {image_seq.dtype}')
number of dimensions: 3
shape: (60, 1440, 1440)
dtype: uint16
資料集是一個影像堆疊,具有 60 個影格(時間點)和 2 個空間維度。讓我們透過每六個時間點採樣來視覺化 10 個影格:我們可以觀察到光照的一些變化。我們利用 Plotly 的 imshow 函數中的 animation_frame 參數。附帶一提,當 binary_string 參數設定為 True 時,影像會以灰階表示。
fig = px.imshow(
image_seq[::6, :, :],
animation_frame=0,
binary_string=True,
labels={'animation_frame': '6-step time point'},
title='Sample of in-vivo human cornea',
)
fig.update_layout(autosize=False, minreducedwidth=250, minreducedheight=250)
plotly.io.show(fig)
隨時間聚合#
首先,我們想要偵測那些資料遺失的污垢點。以技術術語來說,我們想要分割污垢點(針對序列中的所有影格)。與實際資料(訊號)不同,污垢點不會從一個影格移動到下一個影格;它們是靜止的。因此,我們首先計算影像序列的時間聚合。我們應使用中間值影像來分割污垢點,後者隨後相對於背景(模糊訊號)突出顯示。作為補充,為了感受(移動的)資料,讓我們計算變異數。
image_med = np.median(image_seq, axis=0)
image_var = np.var(image_seq, axis=0)
assert image_var.shape == image_med.shape
print(f'shape: {image_med.shape}')
fig, ax = plt.subplots(ncols=2, figsize=(12, 6))
ax[0].imshow(image_med, cmap='gray')
ax[0].set_title('Image median over time')
ax[1].imshow(image_var, cmap='gray')
ax[1].set_title('Image variance over time')
fig.tight_layout()

shape: (1440, 1440)
使用局部閾值處理#
為了分割污垢點,我們使用閾值處理。我們正在使用的影像光照不均勻,這會導致前景和背景的(絕對)強度發生空間變化。例如,一個區域的平均背景強度可能與另一個(遙遠的)區域不同。因此,跨影像計算不同的閾值(每個區域一個)更為合適。這稱為自適應(或局部)閾值處理,與通常對影像中的所有像素採用單個(全域)閾值的閾值處理程序相反。
當從 filters 模組呼叫 threshold_local 函數時,我們可以變更預設的鄰域大小 (block_size),即光照變化的典型大小(像素數),以及 offset(移動鄰域的加權平均值)。讓我們嘗試 block_size 的兩個不同值
讓我們定義一個方便的函數來並排顯示兩個圖表,以便我們更容易比較它們
def plot_comparison(plot1, plot2, title1, title2):
fig, (ax1, ax2) = plt.subplots(ncols=2, figsize=(12, 6), sharex=True, sharey=True)
ax1.imshow(plot1, cmap='gray')
ax1.set_title(title1)
ax2.imshow(plot2, cmap='gray')
ax2.set_title(title2)
plot_comparison(mask_1, mask_2, "block_size = 21", "block_size = 43")

在第二個遮罩中,「污點」似乎更為明顯,也就是使用較大 block_size 值所產生的遮罩。我們注意到,將偏移參數的值從預設的零值增加,會產生更均勻的背景,讓感興趣的物體更明顯地突出。請注意,切換參數值可以讓我們更深入了解所使用的方法,這通常可以使我們更接近期望的結果。

移除細微特徵#
我們使用形態學濾波器來銳化遮罩並聚焦於污點。兩個基本的形態學運算子是膨脹和侵蝕,其中膨脹(或侵蝕)會將像素設定為結構元素(足跡)定義的鄰域中最亮(或最暗)的值。
在這裡,我們使用 morphology 模組中的 diamond 函式來建立菱形足跡。先進行侵蝕再進行膨脹稱為開運算。首先,我們應用開運算濾波器,以移除小物體和細線,同時保留較大物體的形狀和大小。
footprint = ski.morphology.diamond(3)
mask_open = ski.morphology.opening(mask_2, footprint)
plot_comparison(mask_2, mask_open, "mask before", "after opening")

由於「開運算」圖像從侵蝕運算開始,因此小於結構元素的亮區域已被移除。在應用開運算濾波器時,調整足跡參數可讓我們控制移除特徵的細微程度。例如,如果我們在上述內容中使用 footprint = ski.morphology.diamond(1),我們會看到只有較小的特徵會被濾除,因此在遮罩中保留更多的點。相反地,如果我們使用相同半徑的圓盤狀足跡,也就是 footprint = ski.morphology.disk(3),則會濾除更多細微的特徵,因為圓盤的面積大於菱形的面積。
接著,我們可以透過應用膨脹濾波器來加寬偵測到的區域。
mask_dilate = ski.morphology.dilation(mask_open, footprint)
plot_comparison(mask_open, mask_dilate, "Before", "After dilation")
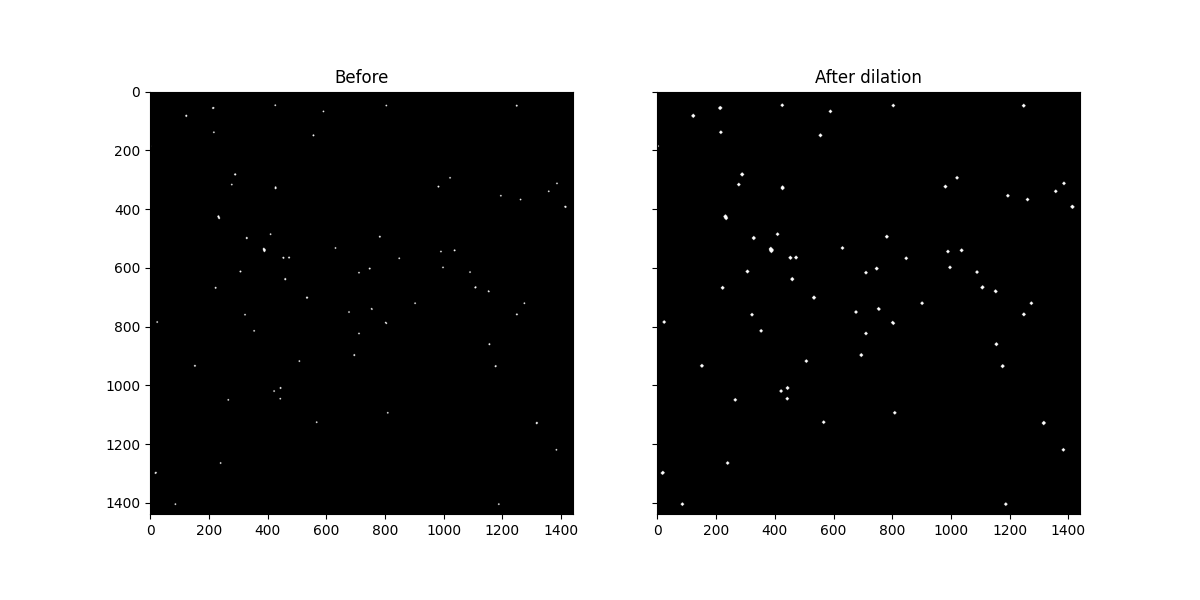
膨脹會擴大亮區域並縮小暗區域。請注意,白點確實是如何加厚的。
分別修復每個影格#
現在我們準備好對每個影格應用修復。為此,我們使用 restoration 模組中的 inpaint_biharmonic 函式。它實作基於雙調和方程式的演算法。此函式採用兩個陣列作為輸入:要修復的影像和對應於我們要修復區域的遮罩(具有相同的形狀)。
image_seq_inpainted = np.zeros(image_seq.shape)
for i in range(image_seq.shape[0]):
image_seq_inpainted[i] = ski.restoration.inpaint_biharmonic(
image_seq[i], mask_dilate
)
讓我們視覺化一個已修復的影像,其中污點已被修復。首先,我們找到污點(或遮罩)的輪廓,以便我們可以將它們繪製在修復的影像之上。
contours = ski.measure.find_contours(mask_dilate)
# Gather all (row, column) coordinates of the contours
x = []
y = []
for contour in contours:
x.append(contour[:, 0])
y.append(contour[:, 1])
# Note that the following one-liner is equivalent to the above:
# x, y = zip(*((contour[:, 0], contour[:, 1]) for contour in contours))
# Flatten the coordinates
x_flat = np.concatenate(x).ravel().round().astype(int)
y_flat = np.concatenate(y).ravel().round().astype(int)
# Create mask of these contours
contour_mask = np.zeros(mask_dilate.shape, dtype=bool)
contour_mask[x_flat, y_flat] = 1
# Pick one frame
sample_result = image_seq_inpainted[12]
# Normalize it (so intensity values range [0, 1])
sample_result /= sample_result.max()
我們使用 color 模組中的 label2rgb 函式,使用透明度(alpha 參數)將修復的影像與分割的點疊加在一起。
color_contours = ski.color.label2rgb(
contour_mask, image=sample_result, alpha=0.4, bg_color=(1, 1, 1)
)
fig, ax = plt.subplots(figsize=(6, 6))
ax.imshow(color_contours)
ax.set_title('Segmented spots over restored image')
fig.tight_layout()
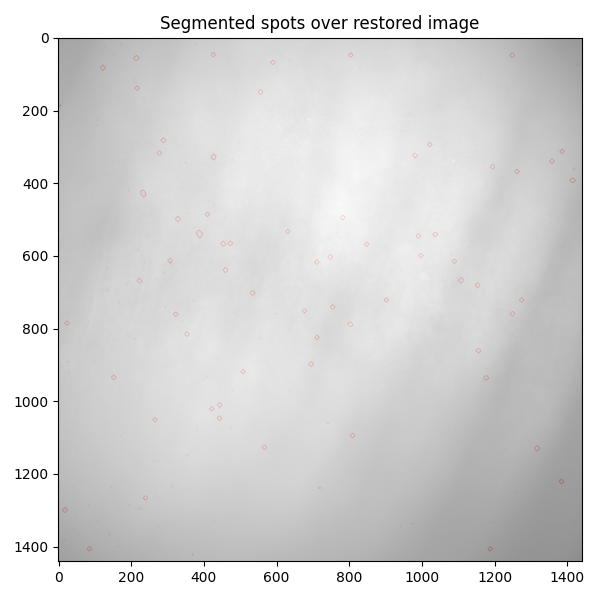
請注意,位於 (x, y) ~ (719, 1237) 的污點很突出;理想情況下,它應該被分割並修復。我們可以發現,在移除細微特徵時,我們在開運算處理步驟中「失去」了它。
plt.show()
腳本總執行時間: (0 分鐘 29.329 秒)
