注意
前往結尾以下載完整的範例程式碼。或者透過 Binder 在您的瀏覽器中執行此範例
骨骼化#
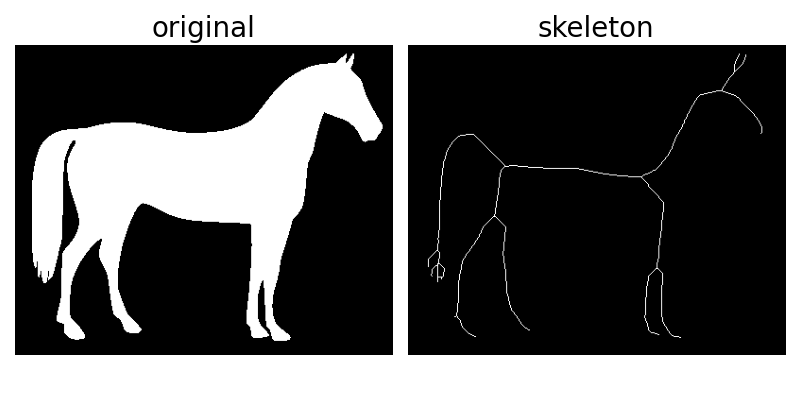
骨骼化將二元物件簡化為 1 個像素寬的表示法。這對於特徵提取和/或表示物件的拓撲結構很有用。
skeletonize 的運作方式是對影像進行連續掃描。在每次掃描中,會識別邊界像素並在它們不破壞相應物件的連通性的條件下移除它們。
from skimage.morphology import skeletonize
from skimage import data
import matplotlib.pyplot as plt
from skimage.util import invert
# Invert the horse image
image = invert(data.horse())
# perform skeletonization
skeleton = skeletonize(image)
# display results
fig, axes = plt.subplots(nrows=1, ncols=2, figsize=(8, 4), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(image, cmap=plt.cm.gray)
ax[0].axis('off')
ax[0].set_title('original', fontsize=20)
ax[1].imshow(skeleton, cmap=plt.cm.gray)
ax[1].axis('off')
ax[1].set_title('skeleton', fontsize=20)
fig.tight_layout()
plt.show()
張氏方法 vs 李氏方法
skeletonize [Zha84] 的運作方式是對影像進行連續掃描,移除物件邊界上的像素。此過程會持續到無法移除更多像素為止。影像會與一個遮罩相關聯,該遮罩會為每個像素分配一個 [0…255] 範圍內的數字,對應於其 8 個相鄰像素的每個可能模式。然後使用查找表為像素分配 0、1、2 或 3 的值,這些值會在迭代期間選擇性地移除。
skeletonize(..., method='lee') [Lee94] 使用八叉樹資料結構來檢查像素的 3x3x3 鄰域。該演算法的進行方式是重複掃描影像,並在每次迭代時移除像素,直到影像停止變化為止。每次迭代包含兩個步驟:首先,組合一個要移除的候選項清單;然後,從此清單中依序重新檢查像素,以更好地保留影像的連通性。
請注意,李氏方法 [Lee94] 設計用於 3D 影像,並且會針對這些影像自動選取。為便於說明,我們將此演算法套用至 2D 影像。
用於細化數位模式的快速並行演算法,T. Y. Zhang 和 C. Y. Suen,《Communications of the ACM》,1984 年 3 月,第 27 卷,第 3 期。
import matplotlib.pyplot as plt
from skimage.morphology import skeletonize
blobs = data.binary_blobs(200, blob_size_fraction=0.2, volume_fraction=0.35, rng=1)
skeleton = skeletonize(blobs)
skeleton_lee = skeletonize(blobs, method='lee')
fig, axes = plt.subplots(1, 3, figsize=(8, 4), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(blobs, cmap=plt.cm.gray)
ax[0].set_title('original')
ax[0].axis('off')
ax[1].imshow(skeleton, cmap=plt.cm.gray)
ax[1].set_title('skeletonize')
ax[1].axis('off')
ax[2].imshow(skeleton_lee, cmap=plt.cm.gray)
ax[2].set_title('skeletonize (Lee 94)')
ax[2].axis('off')
fig.tight_layout()
plt.show()

中間軸骨骼化
物件的中間軸是物件邊界上具有多個最近點的所有點的集合。它通常稱為拓撲骨骼,因為它是物件的 1 像素寬骨骼,與原始物件具有相同的連通性。
在此,我們使用中間軸轉換來計算前景物件的寬度。由於函式 medial_axis 除了中間軸 (使用關鍵字引數 return_distance=True) 之外,還會傳回距離轉換,因此可以使用此函式計算中間軸所有點到背景的距離。這可以估計物件的局部寬度。
對於分支較少的骨骼,應優先使用 skeletonize。
from skimage.morphology import medial_axis, skeletonize
# Generate the data
blobs = data.binary_blobs(200, blob_size_fraction=0.2, volume_fraction=0.35, rng=1)
# Compute the medial axis (skeleton) and the distance transform
skel, distance = medial_axis(blobs, return_distance=True)
# Compare with other skeletonization algorithms
skeleton = skeletonize(blobs)
skeleton_lee = skeletonize(blobs, method='lee')
# Distance to the background for pixels of the skeleton
dist_on_skel = distance * skel
fig, axes = plt.subplots(2, 2, figsize=(8, 8), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(blobs, cmap=plt.cm.gray)
ax[0].set_title('original')
ax[0].axis('off')
ax[1].imshow(dist_on_skel, cmap='magma')
ax[1].contour(blobs, [0.5], colors='w')
ax[1].set_title('medial_axis')
ax[1].axis('off')
ax[2].imshow(skeleton, cmap=plt.cm.gray)
ax[2].set_title('skeletonize')
ax[2].axis('off')
ax[3].imshow(skeleton_lee, cmap=plt.cm.gray)
ax[3].set_title("skeletonize (Lee 94)")
ax[3].axis('off')
fig.tight_layout()
plt.show()

形態細化
在 thin 函式中實作的形態細化,與 skeletonize 的原理相同:在每次迭代中從邊界移除像素,直到無法移除任何像素而不改變連通性為止。不同的移除規則可以加速骨骼化,並產生不同的最終骨骼。
thin 函式還接受可選的 max_num_iter 關鍵字引數,以限制細化迭代的次數,從而產生相對較厚的骨骼。
from skimage.morphology import skeletonize, thin
skeleton = skeletonize(image)
thinned = thin(image)
thinned_partial = thin(image, max_num_iter=25)
fig, axes = plt.subplots(2, 2, figsize=(8, 8), sharex=True, sharey=True)
ax = axes.ravel()
ax[0].imshow(image, cmap=plt.cm.gray)
ax[0].set_title('original')
ax[0].axis('off')
ax[1].imshow(skeleton, cmap=plt.cm.gray)
ax[1].set_title('skeleton')
ax[1].axis('off')
ax[2].imshow(thinned, cmap=plt.cm.gray)
ax[2].set_title('thinned')
ax[2].axis('off')
ax[3].imshow(thinned_partial, cmap=plt.cm.gray)
ax[3].set_title('partially thinned')
ax[3].axis('off')
fig.tight_layout()
plt.show()

腳本的總執行時間: (0 分鐘 2.332 秒)
