注意
前往結尾下載完整的範例程式碼。或透過 Binder 在您的瀏覽器中執行此範例
測量核膜的螢光強度#
此範例重現了生物影像數據分析中一個完善的工作流程,用於測量定位在核膜上的螢光強度,在細胞影像的時間序列(每個影像具有兩個通道和兩個空間維度)中,顯示了蛋白質從細胞質區域重新定位到核膜的過程。這個生物應用程式最初由 Andrea Boni 及其合作者在 [1] 中提出;它被 Kota Miura 在教科書 [2] 以及其他著作 ([3], [4]) 中使用。換句話說,我們將此工作流程從 ImageJ Macro 移植到 Python 與 scikit-image。
import matplotlib.pyplot as plt
import numpy as np
import plotly.io
import plotly.express as px
from scipy import ndimage as ndi
from skimage import filters, measure, morphology, segmentation
from skimage.data import protein_transport
我們先從單個細胞/細胞核開始構建工作流程。
image_sequence = protein_transport()
print(f'shape: {image_sequence.shape}')
shape: (15, 2, 180, 183)
資料集是一個 2D 影像堆疊,包含 15 個影格(時間點)和 2 個通道。
vmin, vmax = 0, image_sequence.max()
fig = px.imshow(
image_sequence,
facet_col=1,
animation_frame=0,
zmin=vmin,
zmax=vmax,
binary_string=True,
labels={'animation_frame': 'time point', 'facet_col': 'channel'},
)
plotly.io.show(fig)
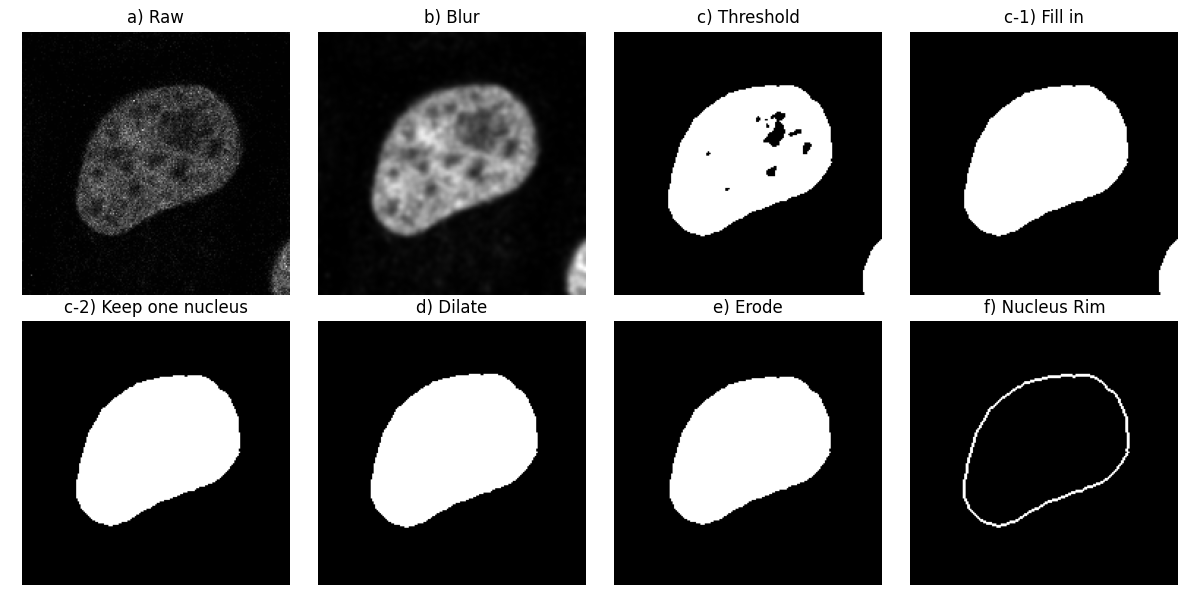
首先,讓我們考慮第一個影像的第一個通道(下圖中的步驟 a))。
image_t_0_channel_0 = image_sequence[0, 0, :, :]
分割細胞核邊緣#
讓我們將高斯低通濾波器應用於此影像以使其平滑(步驟 b))。接下來,我們分割細胞核,使用 Otsu 的方法找到背景和前景之間的閾值:我們得到一個二元影像(步驟 c))。然後,我們填補物件中的孔洞(步驟 c-1))。
smooth = filters.gaussian(image_t_0_channel_0, sigma=1.5)
thresh_value = filters.threshold_otsu(smooth)
thresh = smooth > thresh_value
fill = ndi.binary_fill_holes(thresh)
依照原始工作流程,讓我們移除接觸影像邊界的物件(步驟 c-2))。在這裡,我們可以看見另一個細胞核的一部分接觸到右下角。
dtype('bool')
我們計算此二元影像的形態膨脹(步驟 d))和形態侵蝕(步驟 e))。
最後,我們從膨脹中減去侵蝕以得到細胞核邊緣(步驟 f))。這相當於選擇 dilate 中但不屬於 erode 的像素
mask = np.logical_and(dilate, ~erode)
讓我們在子圖序列中視覺化這些處理步驟。
fig, ax = plt.subplots(2, 4, figsize=(12, 6), sharey=True)
ax[0, 0].imshow(image_t_0_channel_0, cmap=plt.cm.gray)
ax[0, 0].set_title('a) Raw')
ax[0, 1].imshow(smooth, cmap=plt.cm.gray)
ax[0, 1].set_title('b) Blur')
ax[0, 2].imshow(thresh, cmap=plt.cm.gray)
ax[0, 2].set_title('c) Threshold')
ax[0, 3].imshow(fill, cmap=plt.cm.gray)
ax[0, 3].set_title('c-1) Fill in')
ax[1, 0].imshow(clear, cmap=plt.cm.gray)
ax[1, 0].set_title('c-2) Keep one nucleus')
ax[1, 1].imshow(dilate, cmap=plt.cm.gray)
ax[1, 1].set_title('d) Dilate')
ax[1, 2].imshow(erode, cmap=plt.cm.gray)
ax[1, 2].set_title('e) Erode')
ax[1, 3].imshow(mask, cmap=plt.cm.gray)
ax[1, 3].set_title('f) Nucleus Rim')
for a in ax.ravel():
a.set_axis_off()
fig.tight_layout()
將分割的邊緣作為遮罩應用#
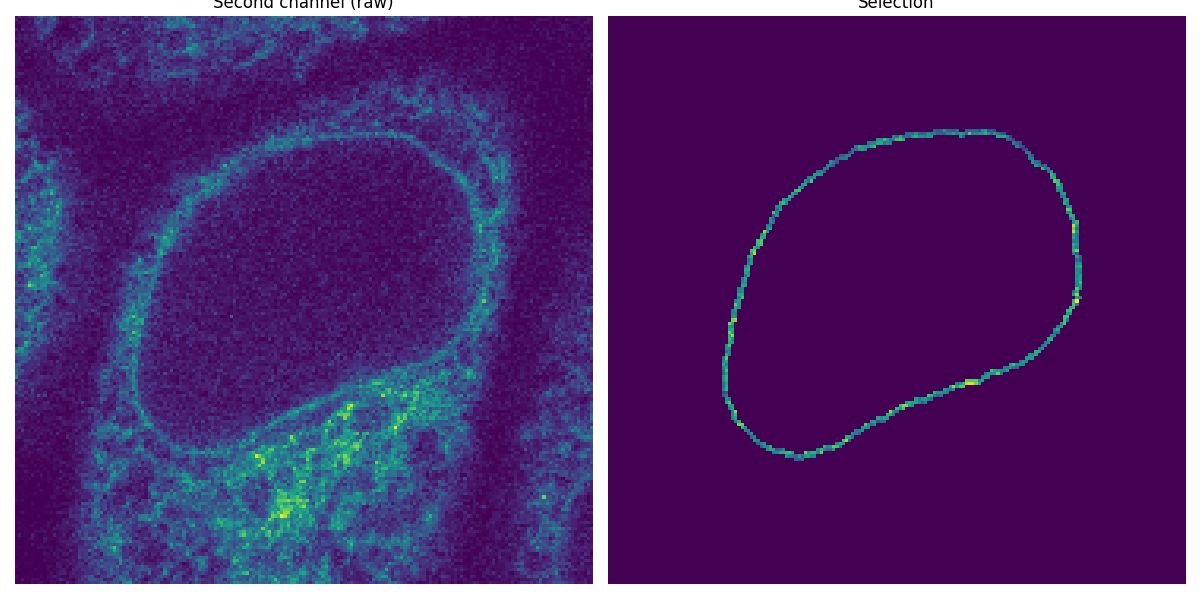
現在我們已經分割了第一個通道中的核膜,我們將其用作遮罩來測量第二個通道中的強度。
image_t_0_channel_1 = image_sequence[0, 1, :, :]
selection = np.where(mask, image_t_0_channel_1, 0)
fig, (ax0, ax1) = plt.subplots(1, 2, figsize=(12, 6), sharey=True)
ax0.imshow(image_t_0_channel_1)
ax0.set_title('Second channel (raw)')
ax0.set_axis_off()
ax1.imshow(selection)
ax1.set_title('Selection')
ax1.set_axis_off()
fig.tight_layout()