skimage.segmentation#
將影像分割成有意義的區域或邊界的演算法。
主動輪廓模型。 |
|
Chan-Vese 分割演算法。 |
|
建立具有二元值的棋盤格水平集。 |
|
清除連接到標籤影像邊界的物件。 |
|
建立具有二元值的圓盤水平集。 |
|
在標籤影像中將標籤擴展 |
|
計算 Felzenszwalb 基於圖形的有效率影像分割。 |
|
傳回布林陣列,其中標籤區域之間的邊界為 True。 |
|
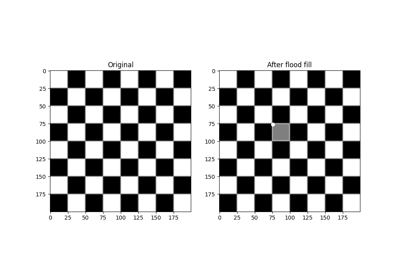
對應於泛洪填充的遮罩。 |
|
對影像執行泛洪填充。 |
|
梯度大小的倒數。 |
|
傳回兩個輸入分割的聯集。 |
|
傳回影像,其中會醒目標示標籤區域之間的邊界。 |
|
無邊緣的形態學主動輪廓 (MorphACWE) |
|
形態學大地主動輪廓 (MorphGAC)。 |
|
在色彩-(x,y) 空間中使用 quickshift 分群來分割影像。 |
|
用於從標記進行分割的隨機遊走演算法。 |
|
將任意標籤重新標記為 { |
|
在色彩-(x,y,z) 空間中使用 k-means 分群來分割影像。 |
|
在從給定標記淹沒的影像中尋找分水嶺盆地。 |
- skimage.segmentation.active_contour(image, snake, alpha=0.01, beta=0.1, w_line=0, w_edge=1, gamma=0.01, max_px_move=1.0, max_num_iter=2500, convergence=0.1, *, boundary_condition='periodic')[原始碼]#
主動輪廓模型。
透過將蛇形擬合至影像的特徵來建立主動輪廓。支援單通道和多通道 2D 影像。蛇形可以是週期性的 (用於分割),或是具有固定和/或自由端點。輸出蛇形的長度與輸入邊界相同。由於點的數量是固定的,請確保初始蛇形具有足夠的點來捕捉最終輪廓的細節。
- 參數:
- image(M, N) 或 (M, N, 3) ndarray
輸入影像。
- snake(K, 2) ndarray
初始蛇形座標。對於週期性邊界條件,端點不得重複。
- alphafloat,選用
蛇形長度形狀參數。值越高,蛇形收縮速度越快。
- betafloat,選用
蛇形平滑度形狀參數。值越高,蛇形越平滑。
- w_linefloat,選用
控制對亮度的吸引力。使用負值吸引朝向黑暗區域。
- w_edgefloat,選用
控制對邊緣的吸引力。使用負值將蛇形從邊緣排斥開。
- gammafloat,選用
明確時間步進參數。
- max_px_movefloat,選用
每次迭代移動的最大像素距離。
- max_num_iterint,選用
最佳化蛇形形狀的最大迭代次數。
- convergencefloat,選用
收斂條件。
- boundary_conditionstring,選用
輪廓的邊界條件。可以是 'periodic'、'free'、'fixed'、'free-fixed' 或 'fixed-free' 其中之一。'periodic' 會附加蛇形的兩個端點,'fixed' 會將端點固定在原位,而 'free' 允許端點自由移動。可以透過剖析 'fixed-free'、'free-fixed' 來組合 'fixed' 和 'free'。剖析 'fixed-fixed' 或 'free-free' 會分別產生與 'fixed' 和 'free' 相同的行為。
- 傳回:
- snake(K, 2) ndarray
最佳化的蛇形,與輸入參數的形狀相同。
參考文獻
[1]Kass, M.; Witkin, A.; Terzopoulos, D. “Snakes: Active contour models”. International Journal of Computer Vision 1 (4): 321 (1988). DOI:10.1007/BF00133570
範例
>>> from skimage.draw import circle_perimeter >>> from skimage.filters import gaussian
建立並平滑影像
>>> img = np.zeros((100, 100)) >>> rr, cc = circle_perimeter(35, 45, 25) >>> img[rr, cc] = 1 >>> img = gaussian(img, sigma=2, preserve_range=False)
初始化樣條
>>> s = np.linspace(0, 2*np.pi, 100) >>> init = 50 * np.array([np.sin(s), np.cos(s)]).T + 50
將樣條擬合至影像
>>> snake = active_contour(img, init, w_edge=0, w_line=1) >>> dist = np.sqrt((45-snake[:, 0])**2 + (35-snake[:, 1])**2) >>> int(np.mean(dist)) 25
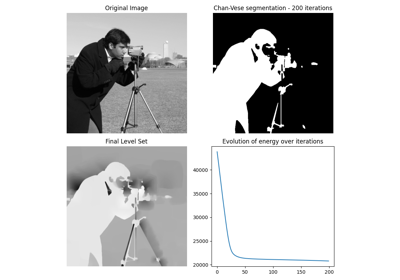
- skimage.segmentation.chan_vese(image, mu=0.25, lambda1=1.0, lambda2=1.0, tol=0.001, max_num_iter=500, dt=0.5, init_level_set='checkerboard', extended_output=False)[原始碼]#
Chan-Vese 分割演算法。
透過演變水平集來建立主動輪廓模型。可用於分割沒有明確定義邊界的物件。
- 參數:
- image(M, N) ndarray
要分割的灰階影像。
- mufloat,選用
「邊緣長度」權重參數。較高的
mu值將產生「圓形」邊緣,而接近零的值將偵測較小的物件。- lambda1float,選用
數值為 ‘True’ 的輸出區域的「與平均值的差異」權重參數。如果它低於
lambda2,則此區域的值範圍將大於另一個區域。- lambda2float,選用
數值為 ‘False’ 的輸出區域的「與平均值的差異」權重參數。如果它低於
lambda1,則此區域的值範圍將大於另一個區域。- tolfloat,正數,選用
迭代之間的水平集變化容差。如果連續迭代的水平集之間的 L2 範數差異,除以影像的面積,低於此值,則演算法會假設已達到解。
- max_num_iteruint,選用
演算法自我中斷前允許的最大迭代次數。
- dtfloat,選用
套用至每個步驟計算的乘法因子,可加速演算法。雖然較高的值可能會加速演算法,但它們也可能導致收斂問題。
- init_level_setstr 或 (M, N) ndarray,選用
定義演算法使用的起始水平集。如果輸入字串,則會自動產生符合影像大小的水平集。或者,可以定義自訂水平集,該水平集應為與 ‘image’ 具有相同形狀的浮點值陣列。接受的字串值如下所示。
- ‘checkerboard’
起始水平集定義為 sin(x/5*pi)*sin(y/5*pi),其中 x 和 y 是像素座標。此水平集具有快速收斂性,但可能無法偵測隱含邊緣。
- ‘disk’
起始水平集定義為與影像中心距離的反向,減去影像寬度和影像高度之間最小值的一半。這速度稍慢,但更有可能正確偵測隱含邊緣。
- ‘small disk’
起始水平集定義為與影像中心距離的反向,減去影像寬度和影像高度之間最小值的四分之一。
- extended_outputbool,選用
如果設定為 True,則傳回值將是一個包含三個傳回值的元組 (請參閱下文)。如果設定為 False,這是預設值,則只會傳回 ‘segmentation’ 陣列。
- 傳回:
- segmentation(M, N) ndarray,bool
演算法產生的分割。
- phi(M, N) 浮點數的 ndarray
演算法計算的最終水平集。
- energies浮點數列表
顯示演算法每個步驟的「能量」演變。這應允許檢查演算法是否收斂。
附註
Chan-Vese 演算法旨在分割邊界不明確的物體。此演算法基於水平集,透過迭代演化來最小化能量,能量由加權值定義,這些加權值對應於分割區域外部的強度與平均值的差異總和、分割區域內部的強度與平均值的差異總和,以及取決於分割區域邊界長度的一項。
此演算法最早由 Tony Chan 和 Luminita Vese 在一篇題為「無邊緣的主動輪廓模型」的論文中提出[1]。
此演算法的實作在某種程度上有所簡化,因為原始論文中描述的面積因子 ‘nu’ 並未實作,且僅適用於灰階影像。
lambda1和lambda2的典型值為 1。如果「背景」在分佈上與分割的物體非常不同(例如,具有不同強度圖案的均勻黑色影像),則這些值應彼此不同。mu 的典型值介於 0 和 1 之間,但處理輪廓不明確的形狀時可以使用較高的值。
此演算法試圖最小化的「能量」定義為區域內與平均值的差異平方的總和,並由 ‘lambda’ 因子加權,再加上輪廓長度乘以 ‘mu’ 因子。
僅支援 2D 灰階影像,且未實作原始文章中描述的面積項。
參考文獻
[1]無邊緣的主動輪廓模型,Tony Chan 和 Luminita Vese,電腦視覺中的尺度空間理論,1999,DOI:10.1007/3-540-48236-9_13
[2]Chan-Vese 分割,Pascal Getreuer 線上影像處理,2 (2012),pp. 214-224,DOI:10.5201/ipol.2012.g-cv
[3]Chan-Vese 演算法 - 專案報告,Rami Cohen,2011 arXiv:1107.2782
- skimage.segmentation.checkerboard_level_set(image_shape, square_size=5)[原始碼]#
建立具有二元值的棋盤格水平集。
- 參數:
- image_shape正整數的 tuple
影像的形狀。
- square_sizeint,選用
棋盤格的正方形大小。預設值為 5。
- 傳回:
- out形狀為
image_shape的陣列 棋盤格的二元水平集。
- out形狀為
- skimage.segmentation.clear_border(labels, buffer_size=0, bgval=0, mask=None, *, out=None)[原始碼]#
清除連接到標籤影像邊界的物件。
- 參數:
- labels(M[, N[, …, P]]) int 或 bool 陣列
影像資料標籤。
- buffer_sizeint,選用
所檢查的邊框寬度。預設情況下,只會移除接觸影像外部的物件。
- bgvalfloat 或 int,選用
清除的物件會設定為此值。
- mask與
image相同形狀的 bool ndarray,選用。 影像資料遮罩。與遮罩的 False 像素重疊的 labels 影像中的物件將會被移除。如果已定義,則會忽略引數 buffer_size。
- outndarray
與
labels相同形狀的陣列,輸出會放置在其中。預設情況下,會建立一個新陣列。
- 傳回:
- out(M[, N[, …, P]]) 陣列
具有清除邊框的影像資料標籤
範例
>>> import numpy as np >>> from skimage.segmentation import clear_border >>> labels = np.array([[0, 0, 0, 0, 0, 0, 0, 1, 0], ... [1, 1, 0, 0, 1, 0, 0, 1, 0], ... [1, 1, 0, 1, 0, 1, 0, 0, 0], ... [0, 0, 0, 1, 1, 1, 1, 0, 0], ... [0, 1, 1, 1, 1, 1, 1, 1, 0], ... [0, 0, 0, 0, 0, 0, 0, 0, 0]]) >>> clear_border(labels) array([[0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 1, 0, 0, 0, 0], [0, 0, 0, 1, 0, 1, 0, 0, 0], [0, 0, 0, 1, 1, 1, 1, 0, 0], [0, 1, 1, 1, 1, 1, 1, 1, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0]]) >>> mask = np.array([[0, 0, 1, 1, 1, 1, 1, 1, 1], ... [0, 0, 1, 1, 1, 1, 1, 1, 1], ... [1, 1, 1, 1, 1, 1, 1, 1, 1], ... [1, 1, 1, 1, 1, 1, 1, 1, 1], ... [1, 1, 1, 1, 1, 1, 1, 1, 1], ... [1, 1, 1, 1, 1, 1, 1, 1, 1]]).astype(bool) >>> clear_border(labels, mask=mask) array([[0, 0, 0, 0, 0, 0, 0, 1, 0], [0, 0, 0, 0, 1, 0, 0, 1, 0], [0, 0, 0, 1, 0, 1, 0, 0, 0], [0, 0, 0, 1, 1, 1, 1, 0, 0], [0, 1, 1, 1, 1, 1, 1, 1, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0]])
- skimage.segmentation.disk_level_set(image_shape, *, center=None, radius=None)[原始碼]#
建立具有二元值的圓盤水平集。
- 參數:
- image_shape正整數的 tuple
影像的形狀
- center正整數的 tuple,選用
磁碟中心點的座標,以 (列、行) 表示。如果未給定,則預設為影像的中心。
- radiusfloat,選用
磁碟的半徑。如果未給定,則會設定為最小影像尺寸的 75%。
- 傳回:
- out形狀為
image_shape的陣列 具有給定
radius和center的磁碟的二元水平集。
- out形狀為
- skimage.segmentation.expand_labels(label_image, distance=1, spacing=1)[原始碼]#
在標籤影像中將標籤擴展
distance個像素,且不重疊。給定標籤影像,
expand_labels會將標籤區域(連通元件)向外擴展最多distance個單位,而不會溢位到相鄰區域中。更具體地說,每個與連通元件的歐幾里得距離 <=distance像素的背景像素都會被指定該連通元件的標籤。spacing參數可用於指定距離轉換的間距率,以計算非等向影像的歐幾里得距離。如果多個連通元件與背景像素的距離在distance像素內,則會指定最近的連通元件的標籤值(請參閱「注意事項」以了解多個標籤距離相等的情況)。- 參數:
- label_imagedtype 為 int 的 ndarray
標籤影像
- distancefloat
以像素為單位擴展標籤的歐幾里得距離。預設值為 1。
- spacingfloat 或 float 序列,選用
沿著每個維度的元素間距。如果是序列,則長度必須等於輸入的秩;如果為單個數字,則會將其用於所有軸。如果未指定,則表示網格間距為 1。
- 傳回:
- enlarged_labelsdtype 為 int 的 ndarray
已標籤陣列,其中所有連通區域都已擴大
附註
如果標籤的間隔距離超過
distance個像素,則這相當於使用半徑為distance的磁碟或超球進行形態學膨脹。但是,與形態學膨脹不同,expand_labels不會將標籤區域擴展到相鄰區域中。expand_labels的這個實作源自 CellProfiler [1],在 CellProfiler 中稱為模組「IdentifySecondaryObjects (Distance-N)」[2]。當一個像素與多個區域的距離相同時,會有一個重要的邊緣情況,因為未定義哪個區域會擴展到該空間中。在這裡,確切的行為取決於
scipy.ndimage.distance_transform_edt的上游實作。參考文獻
範例
>>> labels = np.array([0, 1, 0, 0, 0, 0, 2]) >>> expand_labels(labels, distance=1) array([1, 1, 1, 0, 0, 2, 2])
標籤不會互相覆蓋
>>> expand_labels(labels, distance=3) array([1, 1, 1, 1, 2, 2, 2])
如果出現平手,則行為未定義,但目前會解析為按詞典順序最接近
(0,) * ndim的標籤。>>> labels_tied = np.array([0, 1, 0, 2, 0]) >>> expand_labels(labels_tied, 1) array([1, 1, 1, 2, 2]) >>> labels2d = np.array( ... [[0, 1, 0, 0], ... [2, 0, 0, 0], ... [0, 3, 0, 0]] ... ) >>> expand_labels(labels2d, 1) array([[2, 1, 1, 0], [2, 2, 0, 0], [2, 3, 3, 0]]) >>> expand_labels(labels2d, 1, spacing=[1, 0.5]) array([[1, 1, 1, 1], [2, 2, 2, 0], [3, 3, 3, 3]])
- skimage.segmentation.felzenszwalb(image, scale=1, sigma=0.8, min_size=20, *, channel_axis=-1)[原始碼]#
計算 Felsenszwalb 基於圖形的有效影像分割。
使用在影像網格上基於最小生成樹的快速叢集,產生多通道(即 RGB)影像的過度分割。
scale參數設定觀察級別。較高的 scale 表示較少且較大的區段。sigma是高斯核心的直徑,用於在分割前平滑影像。產生的區塊數量及其大小只能透過
scale間接控制。圖像內的區塊大小可能會因局部對比度而有很大差異。對於 RGB 圖像,該演算法使用色彩空間中像素之間的歐幾里德距離。
- 參數:
- image(M, N[, 3]) ndarray
輸入影像。
- scalefloat
自由參數。值越高表示叢集越大。
- sigmafloat
預處理中使用的 Gaussian 核心的寬度(標準差)。
- min_sizeint
最小元件大小。使用後處理強制執行。
- channel_axisint 或 None,可選
如果為 None,則假定圖像為灰階(單通道)圖像。否則,此參數指示陣列的哪個軸對應於通道。
在版本 0.19 中新增:
channel_axis已在 0.19 中新增。
- 傳回:
- segment_mask(M, N) ndarray
整數遮罩,指示區塊標籤。
附註
原始論文中使用的
k參數在此處重新命名為scale。參考文獻
[1]高效的基於圖形的圖像分割,Felzenszwalb, P.F. 和 Huttenlocher, D.P.,《國際計算機視覺雜誌》,2004
範例
>>> from skimage.segmentation import felzenszwalb >>> from skimage.data import coffee >>> img = coffee() >>> segments = felzenszwalb(img, scale=3.0, sigma=0.95, min_size=5)
- skimage.segmentation.find_boundaries(label_img, connectivity=1, mode='thick', background=0)[source]#
傳回布林陣列,其中標籤區域之間的邊界為 True。
- 參數:
- label_imgint 或 bool 陣列
一個陣列,其中不同的區域標記有不同的整數或布林值。
- connectivityint,在 {1, …,
label_img.ndim} 中,可選 如果任何鄰居具有不同的標籤,則像素被視為邊界像素。
connectivity控制哪些像素被視為鄰居。連通性為 1(預設值)表示共享邊緣(在 2D 中)或面(在 3D 中)的像素將被視為鄰居。連通性為label_img.ndim表示共享角落的像素將被視為鄰居。- mode字串,在 {‘thick’, ‘inner’, ‘outer’, ‘subpixel’} 中
如何標記邊界
thick:任何未被相同標籤的像素完全包圍(由
connectivity定義)的像素都標記為邊界。這會導致邊界為 2 個像素寬。inner:勾勒出物件*內側*的像素,保持背景像素不變。
outer:勾勒出物件邊界周圍背景中的像素。當兩個物件接觸時,也會標記它們的邊界。
subpixel:傳回一個加倍的圖像,其中在原始像素*之間*的像素在適當的地方標記為邊界。
- backgroundint,可選
對於模式 ‘inner’ 和 ‘outer’,需要定義背景標籤。有關這兩個標籤的說明,請參閱
mode。
- 傳回:
- boundaries布林值陣列,與
label_img具有相同的形狀 布林值圖像,其中
True表示邊界像素。對於等於 ‘subpixel’ 的mode,對於所有i,boundaries.shape[i]等於2 * label_img.shape[i] - 1(在所有其他像素對之間插入一個像素)。
- boundaries布林值陣列,與
範例
>>> labels = np.array([[0, 0, 0, 0, 0, 0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0, 0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0, 5, 5, 5, 0, 0], ... [0, 0, 1, 1, 1, 5, 5, 5, 0, 0], ... [0, 0, 1, 1, 1, 5, 5, 5, 0, 0], ... [0, 0, 1, 1, 1, 5, 5, 5, 0, 0], ... [0, 0, 0, 0, 0, 5, 5, 5, 0, 0], ... [0, 0, 0, 0, 0, 0, 0, 0, 0, 0], ... [0, 0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=np.uint8) >>> find_boundaries(labels, mode='thick').astype(np.uint8) array([[0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 1, 1, 1, 0], [0, 1, 1, 1, 1, 1, 0, 1, 1, 0], [0, 1, 1, 0, 1, 1, 0, 1, 1, 0], [0, 1, 1, 1, 1, 1, 0, 1, 1, 0], [0, 0, 1, 1, 1, 1, 1, 1, 1, 0], [0, 0, 0, 0, 0, 1, 1, 1, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=uint8) >>> find_boundaries(labels, mode='inner').astype(np.uint8) array([[0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 0, 1, 0, 0], [0, 0, 1, 0, 1, 1, 0, 1, 0, 0], [0, 0, 1, 1, 1, 1, 0, 1, 0, 0], [0, 0, 0, 0, 0, 1, 1, 1, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=uint8) >>> find_boundaries(labels, mode='outer').astype(np.uint8) array([[0, 0, 0, 0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 0, 0, 1, 1, 1, 0, 0], [0, 0, 1, 1, 1, 1, 0, 0, 1, 0], [0, 1, 0, 0, 1, 1, 0, 0, 1, 0], [0, 1, 0, 0, 1, 1, 0, 0, 1, 0], [0, 1, 0, 0, 1, 1, 0, 0, 1, 0], [0, 0, 1, 1, 1, 1, 0, 0, 1, 0], [0, 0, 0, 0, 0, 1, 1, 1, 0, 0], [0, 0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=uint8) >>> labels_small = labels[::2, ::3] >>> labels_small array([[0, 0, 0, 0], [0, 0, 5, 0], [0, 1, 5, 0], [0, 0, 5, 0], [0, 0, 0, 0]], dtype=uint8) >>> find_boundaries(labels_small, mode='subpixel').astype(np.uint8) array([[0, 0, 0, 0, 0, 0, 0], [0, 0, 0, 1, 1, 1, 0], [0, 0, 0, 1, 0, 1, 0], [0, 1, 1, 1, 0, 1, 0], [0, 1, 0, 1, 0, 1, 0], [0, 1, 1, 1, 0, 1, 0], [0, 0, 0, 1, 0, 1, 0], [0, 0, 0, 1, 1, 1, 0], [0, 0, 0, 0, 0, 0, 0]], dtype=uint8) >>> bool_image = np.array([[False, False, False, False, False], ... [False, False, False, False, False], ... [False, False, True, True, True], ... [False, False, True, True, True], ... [False, False, True, True, True]], ... dtype=bool) >>> find_boundaries(bool_image) array([[False, False, False, False, False], [False, False, True, True, True], [False, True, True, True, True], [False, True, True, False, False], [False, True, True, False, False]])
- skimage.segmentation.flood(image, seed_point, *, footprint=None, connectivity=None, tolerance=None)[source]#
對應於泛洪填充的遮罩。
從特定的
seed_point開始,找到與種子值相等或在tolerance範圍內的連接點。- 參數:
- imagendarray
一個 n 維陣列。
- seed_point元組或整數
在
image中用作洪氾填充起始點的點。如果圖像是一維的,則此點可以給定為整數。- footprintndarray,可選
用於確定每個評估像素的鄰域的足跡(結構元素)。它必須僅包含 1 和 0,並且具有與
image相同的維度數量。如果未給定,則所有相鄰像素都被視為鄰域的一部分(完全連接)。- connectivityint,可選
用於確定每個評估像素的鄰域的數字。與中心平方距離小於或等於
connectivity的相鄰像素被視為鄰居。如果footprint不為 None,則忽略。- tolerancefloat 或 int,可選
如果為 None(預設值),則相鄰值必須嚴格等於
image在seed_point處的初始值。這是最快的。如果給定一個值,則會在每個點進行比較,如果在初始值的容差範圍內,也會被填充(包括在內)。
- 傳回:
- maskndarray
傳回與
image具有相同形狀的布林值陣列,其中與種子點連接並等於(或在容差範圍內)的區域的值為 True。所有其他值為 False。
附註
此操作的概念類比是許多點陣圖形程式中的「油漆桶」工具。此函數僅傳回表示填充的遮罩。
如果出於記憶體原因需要索引而不是遮罩,則使用者可以簡單地在結果上執行
numpy.nonzero,儲存索引,然後捨棄此遮罩。範例
>>> from skimage.morphology import flood >>> image = np.zeros((4, 7), dtype=int) >>> image[1:3, 1:3] = 1 >>> image[3, 0] = 1 >>> image[1:3, 4:6] = 2 >>> image[3, 6] = 3 >>> image array([[0, 0, 0, 0, 0, 0, 0], [0, 1, 1, 0, 2, 2, 0], [0, 1, 1, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
以完全連通性(包括對角線)將連通的 1 填充為 5
>>> mask = flood(image, (1, 1)) >>> image_flooded = image.copy() >>> image_flooded[mask] = 5 >>> image_flooded array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [5, 0, 0, 0, 0, 0, 3]])
將連通的 1 填充為 5,排除對角線點(連通性 1)
>>> mask = flood(image, (1, 1), connectivity=1) >>> image_flooded = image.copy() >>> image_flooded[mask] = 5 >>> image_flooded array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
以容差填充
>>> mask = flood(image, (0, 0), tolerance=1) >>> image_flooded = image.copy() >>> image_flooded[mask] = 5 >>> image_flooded array([[5, 5, 5, 5, 5, 5, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 5, 5, 3]])
- skimage.segmentation.flood_fill(image, seed_point, new_value, *, footprint=None, connectivity=None, tolerance=None, in_place=False)[source]#
對影像執行泛洪填充。
從特定的
seed_point開始,找到與種子值相等或在tolerance範圍內的連接點,然後將其設定為new_value。- 參數:
- imagendarray
一個 n 維陣列。
- seed_point元組或整數
在
image中用作洪氾填充起始點的點。如果圖像是一維的,則此點可以給定為整數。- new_value
image類型 設定整個填充的新值。此值必須與
image的 dtype 一致。- footprintndarray,可選
用於確定每個評估像素的鄰域的足跡(結構元素)。它必須僅包含 1 和 0,並且具有與
image相同的維度數量。如果未給定,則所有相鄰像素都被視為鄰域的一部分(完全連接)。- connectivityint,可選
用於確定每個評估像素的鄰域的數字。與中心平方距離小於或等於
connectivity的相鄰像素被視為鄰居。如果footprint不為 None,則忽略。- tolerancefloat 或 int,可選
如果為 None(預設值),則相鄰值必須嚴格等於
image在seed_point處的值才能被填充。這是最快的。如果提供容差,則會填充與種子點相差在正負容差範圍內的相鄰點(包括在內)。- in_placebool,可選
如果為 True,則將洪氾填充直接應用於
image。如果為 False,則傳回洪氾填充的結果,而不會修改輸入的image(預設值)。
- 傳回:
- filledndarray
傳回與
image具有相同形狀的陣列,其中與種子點連接並等於(或在容差範圍內)的區域中的值會被取代為new_value。
附註
此操作的概念類比是許多點陣圖形程式中的「油漆桶」工具。
範例
>>> from skimage.morphology import flood_fill >>> image = np.zeros((4, 7), dtype=int) >>> image[1:3, 1:3] = 1 >>> image[3, 0] = 1 >>> image[1:3, 4:6] = 2 >>> image[3, 6] = 3 >>> image array([[0, 0, 0, 0, 0, 0, 0], [0, 1, 1, 0, 2, 2, 0], [0, 1, 1, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
以完全連通性(包括對角線)將連通的 1 填充為 5
>>> flood_fill(image, (1, 1), 5) array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [5, 0, 0, 0, 0, 0, 3]])
將連通的 1 填充為 5,排除對角線點(連通性 1)
>>> flood_fill(image, (1, 1), 5, connectivity=1) array([[0, 0, 0, 0, 0, 0, 0], [0, 5, 5, 0, 2, 2, 0], [0, 5, 5, 0, 2, 2, 0], [1, 0, 0, 0, 0, 0, 3]])
以容差填充
>>> flood_fill(image, (0, 0), 5, tolerance=1) array([[5, 5, 5, 5, 5, 5, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 2, 2, 5], [5, 5, 5, 5, 5, 5, 3]])
- skimage.segmentation.inverse_gaussian_gradient(image, alpha=100.0, sigma=5.0)[原始碼]#
梯度大小的倒數。
計算影像中梯度的大小,然後將結果反轉到 [0, 1] 的範圍。平坦區域會被賦予接近 1 的值,而接近邊界的區域則被賦予接近 0 的值。
在呼叫
morphological_geodesic_active_contour之前,應將此函數或使用者定義的類似函數應用於影像作為預處理步驟。- 參數:
- image(M, N) 或 (L, M, N) 陣列
灰階影像或體積資料。
- alphafloat,選用
控制反轉的陡峭程度。較大的值會使平坦區域和邊界區域之間的過渡在結果陣列中更陡峭。
- sigmafloat, 選用
應用於影像的高斯濾波器的標準差。
- 傳回:
- gimage(M, N) 或 (L, M, N) 陣列
適用於
morphological_geodesic_active_contour的預處理影像(或體積資料)。
- skimage.segmentation.join_segmentations(s1, s2, return_mapping: bool = False)[原始碼]#
傳回兩個輸入分割的聯集。
S1 和 S2 的聯集 J 定義為:如果兩個體素在 S1 和 S2 中都位於同一個區段,則它們在聯集中也位於同一個區段。
- 參數:
- s1, s2numpy 陣列
s1 和 s2 是形狀相同的標籤欄位。
- return_mappingbool, 選用
如果為 true,則傳回聯集分割標籤到原始標籤的映射。
- 傳回:
- jnumpy 陣列
s1 和 s2 的聯集分割結果。
- map_j_to_s1ArrayMap, 選用
從聯集分割 j 的標籤到 s1 的標籤的映射。
- map_j_to_s2ArrayMap, 選用
從聯集分割 j 的標籤到 s2 的標籤的映射。
範例
>>> from skimage.segmentation import join_segmentations >>> s1 = np.array([[0, 0, 1, 1], ... [0, 2, 1, 1], ... [2, 2, 2, 1]]) >>> s2 = np.array([[0, 1, 1, 0], ... [0, 1, 1, 0], ... [0, 1, 1, 1]]) >>> join_segmentations(s1, s2) array([[0, 1, 3, 2], [0, 5, 3, 2], [4, 5, 5, 3]]) >>> j, m1, m2 = join_segmentations(s1, s2, return_mapping=True) >>> m1 ArrayMap(array([0, 1, 2, 3, 4, 5]), array([0, 0, 1, 1, 2, 2])) >>> np.all(m1[j] == s1) True >>> np.all(m2[j] == s2) True
- skimage.segmentation.mark_boundaries(image, label_img, color=(1, 1, 0), outline_color=None, mode='outer', background_label=0)[原始碼]#
傳回影像,其中會醒目標示標籤區域之間的邊界。
- 參數:
- image(M, N[, 3]) 陣列
灰階或 RGB 影像。
- label_img(M, N) 整數陣列
標籤陣列,其中區域以不同的整數值標記。
- color長度為 3 的序列, 選用
輸出影像中邊界的 RGB 顏色。
- outline_color長度為 3 的序列, 選用
輸出影像中邊界周圍的 RGB 顏色。如果為 None,則不繪製外框。
- mode字串,在 {‘thick’, ‘inner’, ‘outer’, ‘subpixel’} 中, 選用
尋找邊界的模式。
- background_labelint, 選用
要將哪個標籤視為背景(這僅適用於
inner和outer模式)。
- 傳回:
- marked(M, N, 3) 浮點數陣列
一個影像,其中標籤之間的邊界疊加在原始影像上。
- skimage.segmentation.morphological_chan_vese(image, num_iter, init_level_set='checkerboard', smoothing=1, lambda1=1, lambda2=1, iter_callback=<function <lambda>>)[原始碼]#
無邊緣的形態學主動輪廓 (MorphACWE)
使用形態運算子實作的無邊界活動輪廓。它可用於分割影像和體積資料中邊界不明確的物件。要求物件內部平均看起來與外部不同(即,物件的內部區域平均應比外部區域更暗或更亮)。
- 參數:
- image(M, N) 或 (L, M, N) 陣列
要分割的灰階影像或體積資料。
- num_iteruint
要運行的 num_iter 次數
- init_level_set字串、(M, N) 陣列或 (L, M, N) 陣列
初始水平集。如果給定一個陣列,它將被二值化並用作初始水平集。如果給定一個字串,它會定義一個方法來產生與
image形狀合理的初始水平集。可接受的值為 ‘checkerboard’ 和 ‘disk’。請參閱checkerboard_level_set和disk_level_set的文件,以了解有關如何建立這些水平集的詳細資訊。- smoothinguint, 選用
每次迭代套用平滑運算子的次數。合理的值約為 1-4。較大的值會導致更平滑的分割。
- lambda1float,選用
外部區域的權重參數。如果
lambda1大於lambda2,則外部區域將包含比內部區域更大的值範圍。- lambda2float,選用
內部區域的權重參數。如果
lambda2大於lambda1,則內部區域將包含比外部區域更大的值範圍。- iter_callback函式, 選用
如果給定,則此函式在每次迭代時會被呼叫一次,並將目前的水平集作為唯一引數。這對於除錯或在演化過程中繪製中間結果很有用。
- 傳回:
- out(M, N) 或 (L, M, N) 陣列
最終分割結果(即,最終的水平集)
附註
這是 Chan-Vese 演算法的一個版本,它使用形態運算子而不是求解偏微分方程式 (PDE) 來進行輪廓的演化。在此演算法中使用的一組形態運算子被證明與 Chan-Vese PDE 無限接近等效(請參閱 [1])。但是,形態運算子不會像 PDE 中常見的那樣出現數值穩定性問題(不需要為演化找到正確的時間步長),而且計算速度更快。
該演算法及其理論推導在 [1] 中描述。
參考文獻
[1] (1,2)A Morphological Approach to Curvature-based Evolution of Curves and Surfaces, Pablo Márquez-Neila, Luis Baumela, Luis Álvarez. In IEEE Transactions on Pattern Analysis and Machine Intelligence (PAMI), 2014, DOI:10.1109/TPAMI.2013.106
- skimage.segmentation.morphological_geodesic_active_contour(gimage, num_iter, init_level_set='disk', smoothing=1, threshold='auto', balloon=0, iter_callback=<function <lambda>>)[原始碼]#
形態學大地主動輪廓 (MorphGAC)。
使用形態學運算子實現的測地主動輪廓。它可用於分割具有可見但雜亂、凌亂、斷裂邊界的物件。
- 參數:
- gimage(M, N) 或 (L, M, N) 陣列
要分割的預處理影像或體積。這很少是原始影像。相反地,這通常是原始影像的預處理版本,可增強並突顯要分割的物件的邊界(或其他結構)。
morphological_geodesic_active_contour()會嘗試在gimage值較小的區域停止輪廓演化。請參閱inverse_gaussian_gradient()作為執行此預處理的範例函式。請注意,morphological_geodesic_active_contour()的品質可能很大程度地取決於此預處理。- num_iteruint
要執行的 num_iter 次數。
- init_level_set字串、(M, N) 陣列或 (L, M, N) 陣列
初始水平集。如果給定一個陣列,它將被二值化並用作初始水平集。如果給定一個字串,它會定義一個方法來產生與
image形狀合理的初始水平集。可接受的值為 ‘checkerboard’ 和 ‘disk’。請參閱checkerboard_level_set和disk_level_set的文件,以了解有關如何建立這些水平集的詳細資訊。- smoothinguint, 選用
每次迭代套用平滑運算子的次數。合理的值約為 1-4。較大的值會導致更平滑的分割。
- thresholdfloat,選用
影像中值小於此閾值的區域將被視為邊界。輪廓的演化將在這些區域停止。
- balloonfloat,選用
氣球力,用於引導輪廓在影像的非資訊區域,即影像梯度太小而無法將輪廓推向邊界的區域。負值會縮小輪廓,而正值會擴大這些區域中的輪廓。將其設定為零將停用氣球力。
- iter_callback函式, 選用
如果給定,則此函式在每次迭代時會被呼叫一次,並將目前的水平集作為唯一引數。這對於除錯或在演化過程中繪製中間結果很有用。
- 傳回:
- out(M, N) 或 (L, M, N) 陣列
最終分割結果(即,最終的水平集)
附註
這是測地主動輪廓 (Geodesic Active Contours, GAC) 演算法的版本,它使用形態學運算子,而不是求解偏微分方程式 (Partial Differential Equations, PDEs) 來進行輪廓的演化。此演算法中使用的一組形態學運算子被證明在無限小程度上等同於 GAC PDE(請參閱 [1])。但是,形態學運算子不會遭受 PDE 中常見的數值穩定性問題(例如,沒有必要為演化找到正確的時間步長),並且計算速度更快。
演算法及其理論推導在 [1] 中描述。
參考文獻
[1] (1,2)A Morphological Approach to Curvature-based Evolution of Curves and Surfaces, Pablo Márquez-Neila, Luis Baumela, Luis Álvarez. In IEEE Transactions on Pattern Analysis and Machine Intelligence (PAMI), 2014, DOI:10.1109/TPAMI.2013.106
- skimage.segmentation.quickshift(image, ratio=1.0, kernel_size=5, max_dist=10, return_tree=False, sigma=0, convert2lab=True, rng=42, *, channel_axis=-1)[原始碼]#
在色彩-(x,y) 空間中使用 quickshift 分群來分割影像。
使用快速偏移模式搜尋演算法產生影像的過度分割。
- 參數:
- image(M, N, C) ndarray
輸入影像。對應於色彩通道的軸可以透過
channel_axis引數指定。- ratiofloat,選用,介於 0 和 1 之間
平衡色彩空間鄰近度和影像空間鄰近度。較高的值會更重視色彩空間。
- kernel_sizefloat,選用
用於平滑樣本密度的 Gaussian 核心寬度。較高表示叢集較少。
- max_distfloat,選用
資料距離的截斷點。較高表示叢集較少。
- return_treebool,選用
是否傳回完整的分割階層樹和距離。
- sigmafloat, 選用
用於作為預處理的 Gaussian 平滑寬度。零表示不進行平滑。
- convert2labbool,選用
在分割之前是否應將輸入轉換為 Lab 色彩空間。為此,假設輸入為 RGB。
- rng{
numpy.random.Generator, int},選用 虛擬隨機數字產生器。預設情況下,會使用 PCG64 產生器(請參閱
numpy.random.default_rng())。如果rng是整數,則會用於播種產生器。PRNG 用於打破平手,預設情況下會以 42 播種。
- channel_axisint,選用
image中對應於色彩通道的軸。預設為最後一個軸。
- 傳回:
- segment_mask(M, N) ndarray
整數遮罩,指示區塊標籤。
附註
作者建議在分割之前將影像轉換為 Lab 色彩空間,儘管這並非絕對必要。為使其運作,影像必須以 RGB 格式給定。
參考文獻
[1]快速偏移和用於模式搜尋的核方法,Vedaldi, A. 和 Soatto, S.,歐洲電腦視覺會議,2008
- skimage.segmentation.random_walker(data, labels, beta=130, mode='cg_j', tol=0.001, copy=True, return_full_prob=False, spacing=None, *, prob_tol=0.001, channel_axis=None)[原始碼]#
用於從標記進行分割的隨機遊走演算法。
針對灰階或多通道影像實作隨機遊走演算法。
- 參數:
- data(M, N[, P][, C]) ndarray
要以不同階段分割的影像。灰階
data可以是二維或三維的;多通道資料可以是三維或四維的,其中channel_axis指定包含通道的維度。除非使用spacing關鍵字引數,否則會假設資料間距是等向性的。- labels(M, N[, P]) 整數陣列
以不同正整數標記不同階段的種子標記陣列。標記為零的像素是未標記的像素。負標籤對應於不活動的像素,這些像素不會被考慮在內(它們會從圖形中移除)。如果標籤不是連續整數,則會轉換標籤陣列,使標籤成為連續的。在多通道情況下,
labels應具有與data的單一通道相同的形狀,即不含表示通道的最後一個維度。- betafloat,選用
隨機遊走運動的懲罰係數(
beta越大,擴散越困難)。- mode字串,可用選項 {'cg'、'cg_j'、'cg_mg'、'bf'}
用於在隨機遊走演算法中求解線性系統的模式。
‘bf’(暴力法):計算 Laplacian 的 LU 分解。這對於小型影像(<1024x1024)而言速度很快,但對於大型影像(例如,3-D 體積)而言,速度非常慢且耗用大量記憶體。
‘cg’(共軛梯度法):使用 scipy.sparse.linalg 中的共軛梯度法迭代求解線性系統。對於大型影像,此方法比暴力法耗用更少的記憶體,但速度相當慢。
‘cg_j’(帶 Jacobi 前處理器的共軛梯度法):在共軛梯度法迭代期間會套用 Jacobi 前處理器。這可能會加速 ‘cg’ 方法的收斂。
‘cg_mg’(使用多重網格預處理器的共軛梯度法):使用多重網格解算器計算預處理器,然後使用共軛梯度法計算解。此模式需要安裝 pyamg 模組。
- tolfloat,可選
使用基於共軛梯度的方法('cg'、'cg_j' 和 'cg_mg')解線性系統時要達到的容差。
- copybool,可選
如果 copy 為 False,則
labels陣列將被分割的結果覆寫。如果您想節省記憶體,請使用 copy=False。- return_full_probbool,可選
如果為 True,將返回像素屬於每個標籤的機率,而不僅僅是最有可能的標籤。
- spacing浮點數的可迭代物件,可選
每個空間維度中體素之間的間距。如果為
None,則假設每個維度中像素/體素之間的間距為 1。- prob_tolfloat,可選
結果機率在 [0, 1] 區間內的容差。如果容差不滿足,則會顯示警告。
- channel_axisint 或 None,可選
如果為 None,則假定圖像為灰階(單通道)圖像。否則,此參數指示陣列的哪個軸對應於通道。
在版本 0.19 中新增:
channel_axis已在 0.19 中新增。
- 傳回:
- outputndarray
如果
return_full_prob為 False,則會傳回與labels具有相同形狀和資料類型的整數陣列,其中每個像素都根據各向異性擴散首先到達該像素的標記進行標記。如果
return_full_prob為 True,則會傳回形狀為(nlabels, labels.shape)的浮點數陣列。output[label_nb, i, j]是標籤label_nb首先到達像素(i, j)的機率。
參見
skimage.segmentation.watershed一種基於數學形態學和從標記「淹沒」區域的分割演算法。
附註
多通道輸入會使用所有通道數據組合進行縮放。請確保在執行此演算法之前,所有通道都已單獨正規化。
spacing引數專門用於各向異性資料集,其中資料點在一個或多個空間維度上的間距不同。各向異性資料在醫學影像中很常見。該演算法最初在 [1] 中提出。
該演算法針對每個相位的標記上的源,求解無限時間的擴散方程式。像素會以具有最大機率首先擴散到該像素的相位進行標記。
透過最小化每個相位的 x.T L x 來求解擴散方程式,其中 L 是影像加權圖的拉普拉斯算子,而 x 是給定相位的標記透過擴散首先到達像素的機率(x=1 在該相位的標記上,x=0 在其他標記上,並尋找其他係數)。每個像素都被歸屬於其 x 值最大的標籤。影像的拉普拉斯算子 L 定義為
L_ii = d_i,像素 i 的鄰居數量(i 的度數)
L_ij = -w_ij,如果 i 和 j 是相鄰像素
權重 w_ij 是局部梯度範數的遞減函數。這確保了相似值的像素之間更容易擴散。
當拉普拉斯算子被分解為標記像素和未標記像素的塊時
L = M B.T B A
其中第一個索引對應於標記像素,然後對應於未標記像素,最小化一個相位的 x.T L x 等同於求解
A x = - B x_m
其中 x_m = 1 在給定相位的標記上,0 在其他標記上。此線性系統在演算法中使用直接方法求解小影像,並使用迭代方法求解較大的影像。
參考文獻
[1]Leo Grady,Random walks for image segmentation,IEEE Trans Pattern Anal Mach Intell. 2006 Nov;28(11):1768-83. DOI:10.1109/TPAMI.2006.233。
範例
>>> rng = np.random.default_rng() >>> a = np.zeros((10, 10)) + 0.2 * rng.random((10, 10)) >>> a[5:8, 5:8] += 1 >>> b = np.zeros_like(a, dtype=np.int32) >>> b[3, 3] = 1 # Marker for first phase >>> b[6, 6] = 2 # Marker for second phase >>> random_walker(a, b) array([[1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 2, 2, 2, 1, 1], [1, 1, 1, 1, 1, 2, 2, 2, 1, 1], [1, 1, 1, 1, 1, 2, 2, 2, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1], [1, 1, 1, 1, 1, 1, 1, 1, 1, 1]], dtype=int32)
- skimage.segmentation.relabel_sequential(label_field, offset=1)[來源]#
將任意標籤重新標記為 {
offset, …offset+ 標籤數}。此函數還會傳回正向映射(將原始標籤映射到縮減的標籤)和反向映射(將縮減的標籤映射回原始標籤)。
- 參數:
- label_fieldint 的 numpy 陣列,任意形狀
一個標籤陣列,必須是非負整數。
- offsetint,可選
傳回的標籤將從
offset開始,該值應為嚴格正數。
- 傳回:
- relabeledint 的 numpy 陣列,與
label_field具有相同形狀 具有映射到 {offset, …, number_of_labels + offset - 1} 的標籤的輸入標籤欄位。資料類型將與
label_field相同,除非 offset + number_of_labels 導致目前的資料類型溢位。- forward_mapArrayMap
從原始標籤空間到傳回標籤空間的映射。可用於重新套用相同的映射。有關使用方法,請參閱範例。輸出資料類型將與
relabeled相同。- inverse_mapArrayMap
從新標籤空間到原始空間的映射。這可用於從重新標記的標籤欄位重建原始標籤欄位。輸出資料類型將與
label_field相同。
- relabeledint 的 numpy 陣列,與
附註
假設標籤 0 表示背景,並且永遠不會重新映射。
對於某些輸入,正向映射可能會非常大,因為其長度由標籤欄位的最大值給定。但是,在大多數情況下,
label_field.max()比label_field.size小得多,在這些情況下,保證正向映射小於輸入或輸出影像。範例
>>> from skimage.segmentation import relabel_sequential >>> label_field = np.array([1, 1, 5, 5, 8, 99, 42]) >>> relab, fw, inv = relabel_sequential(label_field) >>> relab array([1, 1, 2, 2, 3, 5, 4]) >>> print(fw) ArrayMap: 1 → 1 5 → 2 8 → 3 42 → 4 99 → 5 >>> np.array(fw) array([0, 1, 0, 0, 0, 2, 0, 0, 3, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 4, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 5]) >>> np.array(inv) array([ 0, 1, 5, 8, 42, 99]) >>> (fw[label_field] == relab).all() True >>> (inv[relab] == label_field).all() True >>> relab, fw, inv = relabel_sequential(label_field, offset=5) >>> relab array([5, 5, 6, 6, 7, 9, 8])
- skimage.segmentation.slic(image, n_segments=100, compactness=10.0, max_num_iter=10, sigma=0, spacing=None, convert2lab=None, enforce_connectivity=True, min_size_factor=0.5, max_size_factor=3, slic_zero=False, start_label=1, mask=None, *, channel_axis=-1)[來源]#
在色彩-(x,y,z) 空間中使用 k-means 分群來分割影像。
- 參數:
- image(M, N[, P][, C]) ndarray
輸入影像。可以是 2D 或 3D,以及灰階或多通道(請參閱
channel_axis參數)。輸入影像必須是不含 NaN 的,否則必須遮罩 NaN。- n_segmentsint,可選
分割輸出影像中的(近似)標籤數。
- compactnessfloat,可選
平衡色彩接近度和空間接近度。較高的值會更重視空間接近度,使超像素形狀更趨近正方形/立方體。在 SLICO 模式下,這是初始緊密度。此參數強烈取決於影像對比度和影像中物件的形狀。我們建議在對數刻度上探索可能的值,例如,0.01、0.1、1、10、100,然後在選定的值附近進行微調。
- max_num_iterint,選用
k 平均演算法的最大迭代次數。
- sigmafloat 或浮點數的類陣列物件,可選
用於預處理影像每個維度的 Gaussian 平滑核心的寬度。如果為純量值,則將相同的 sigma 套用於每個維度。零表示不平滑。請注意,如果
sigma是純量且提供了手動體素間距,則會自動縮放(請參閱「注意事項」部分)。如果 sigma 是類陣列物件,則其大小必須符合image的空間維度數量。- spacing浮點數的類陣列 (array-like),可選
沿著每個空間維度的體素間距。預設情況下,
slic假設均勻間距(每個空間維度上的體素解析度相同)。此參數控制 k-means 分群期間沿空間維度的距離權重。- convert2labbool,選用
是否應在分割之前將輸入轉換為 Lab 色彩空間。輸入影像必須為 RGB。 強烈建議使用。當
channel_axis` 不是 None 且 ``image.shape[-1] == 3時,此選項預設為True。- enforce_connectivity布林值 (bool),可選
是否產生連通的區段
- min_size_factor浮點數 (float),可選
要相對於假設的區段大小
`depth*width*height/n_segments`移除的最小區段大小的比例- max_size_factor浮點數 (float),可選
最大連通區段大小的比例。在大多數情況下,值為 3 即可。
- slic_zero布林值 (bool),可選
執行 SLIC-zero,即 SLIC 的零參數模式。 [2]
- start_label整數 (int),可選
標籤的索引起始值。應為 0 或 1。
在 0.17 版本中新增:
start_label在 0.17 版本中引入- maskndarray,可選
如果提供,則僅在 mask 為 True 的位置計算超像素,並使用 k-means 分群策略將種子點均勻分佈在遮罩上。遮罩的維度數必須等於影像的空間維度數。
在 0.17 版本中新增:
mask在 0.17 版本中引入- channel_axisint 或 None,可選
如果為 None,則假定圖像為灰階(單通道)圖像。否則,此參數指示陣列的哪個軸對應於通道。
在版本 0.19 中新增:
channel_axis已在 0.19 中新增。
- 傳回:
- labels2D 或 3D 陣列
整數遮罩,指示區塊標籤。
- 引發:
- ValueError
如果
convert2lab設定為True,但最後一個陣列維度的長度不是 3。- ValueError
如果
start_label不是 0 或 1。- ValueError
如果
image包含未遮罩的 NaN 值。- ValueError
如果
image包含未遮罩的無限值。- ValueError
如果
image是 2D 的,但channel_axis是 -1(預設值)。
附註
如果
sigma > 0,則在分割之前使用高斯核心平滑影像。如果
sigma是純量,且提供了spacing,則核心寬度沿著每個維度除以間距。例如,如果sigma=1且spacing=[5, 1, 1],則有效的sigma為[0.2, 1, 1]。這可確保異向影像的合理平滑。影像會在處理前重新縮放至 [0, 1] 範圍內(忽略遮罩值)。
形狀為 (M, N, 3) 的影像預設會被解譯為 2D RGB 影像。若要將它們解譯為最後一個維度長度為 3 的 3D 影像,請使用
channel_axis=None。引入
start_label是為了處理問題 [4]。標籤索引預設從 1 開始。
參考文獻
[1]Radhakrishna Achanta, Appu Shaji, Kevin Smith, Aurelien Lucchi, Pascal Fua, 和 Sabine Süsstrunk,《SLIC Superpixels Compared to State-of-the-art Superpixel Methods》,TPAMI,2012 年 5 月。 DOI:10.1109/TPAMI.2012.120
[3]Irving, Benjamin. 《maskSLIC: regional superpixel generation with application to local pathology characterisation in medical images.》,2016, arXiv:1606.09518
範例
>>> from skimage.segmentation import slic >>> from skimage.data import astronaut >>> img = astronaut() >>> segments = slic(img, n_segments=100, compactness=10)
增加緊密度參數會產生更多正方形區域
>>> segments = slic(img, n_segments=100, compactness=20)
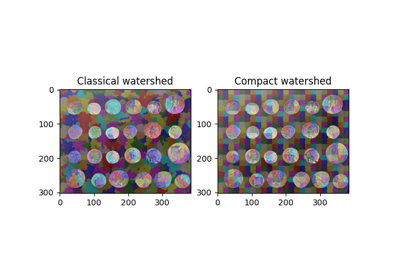
- skimage.segmentation.watershed(image, markers=None, connectivity=1, offset=None, mask=None, compactness=0, watershed_line=False)[原始碼]#
在從給定標記淹沒的影像中尋找分水嶺盆地。
- 參數:
- image(M, N[, …]) ndarray
資料陣列,其中數值最低的點會先標記。
- markers整數 (int),或整數的 (M, N[, …]) ndarray,可選
所需的盆地數量,或是在標籤矩陣中標記盆地要賦予的值的陣列。零表示不是標記。如果為 None,則(預設)標記會判斷為
image的局部最小值。具體而言,計算相當於將skimage.morphology.local_minima()應用於image,然後將skimage.measure.label()應用於結果(使用相同的connectivity)。一般來說,建議使用者明確傳遞標記。- connectivity整數 (int) 或 ndarray,可選
鄰域連通性。整數會按照
scipy.ndimage.generate_binary_structure的方式解譯,表示要到達鄰居所採取的最大正交步驟數。陣列會直接解譯為足跡(結構元素)。預設值為 1。在 2D 中,1 會產生 4 鄰域,而 2 會產生 8 鄰域。- offset類陣列 (array_like),形狀為 image.ndim,可選
足跡中心的座標。
- mask(M, N[, …]) 布林值或 0 和 1 的 ndarray,可選
與
image具有相同形狀的陣列。只有在 mask == True 的點會被標記。- compactnessfloat,可選
使用具有指定緊密度參數的緊湊分水嶺 [1]。較高的值會產生形狀更規則的分水嶺盆地。
- watershed_line布林值 (bool),可選
如果為 True,則一像素寬的線會分隔分水嶺演算法取得的區域。此線的標籤為 0。請注意,用於新增此線的方法假設標記區域不相鄰;分水嶺線可能無法捕捉到相鄰標記區域之間的邊界。
- 傳回:
- outndarray
與
markers具有相同類型和形狀的已標記矩陣。
參見
skimage.segmentation.random_walker一種基於異向擴散的分割演算法,通常比分水嶺慢,但在雜訊資料和有孔洞的邊界上具有良好的結果。
附註
此函式實作了將像素分配到標記盆地的分水嶺演算法 [2] [3]。此演算法使用優先順序佇列來保存像素,而優先順序佇列的度量為像素值,然後是進入佇列的時間 – 這會解決優先順序相同的情況,並偏向最接近的標記。
某些概念取自 [4]。論文中最重要的一個見解是,進入佇列的時間解決了兩個問題:像素應分配給具有最大梯度的鄰居,或者,如果沒有梯度,則平台上的像素應在相對兩側的標記之間分割。
此實作會將所有引數轉換為特定的、最小公分母類型,然後將這些傳遞至 C 演算法。
標記可以手動判斷,或自動判斷,例如使用影像梯度的局部最小值,或使用與背景距離函數的局部最大值來分隔重疊的物件(請參閱範例)。
參考文獻
[1]P. Neubert 和 P. Protzel,「Compact Watershed and Preemptive SLIC: On Improving Trade-offs of Superpixel Segmentation Algorithms」,2014 年第 22 屆國際模式辨識會議,瑞典斯德哥爾摩,2014 年,第 996-1001 頁,DOI:10.1109/ICPR.2014.181 https://www.tu-chemnitz.de/etit/proaut/publications/cws_pSLIC_ICPR.pdf
[4]P. J. Soille 和 M. M. Ansoult,「Automated basin delineation from digital elevation models using mathematical morphology」,Signal Processing,20(2):171-182,DOI:10.1016/0165-1684(90)90127-K
範例
分水嶺演算法可用於分隔重疊的物件。
我們首先產生兩個重疊圓形的初始影像
>>> x, y = np.indices((80, 80)) >>> x1, y1, x2, y2 = 28, 28, 44, 52 >>> r1, r2 = 16, 20 >>> mask_circle1 = (x - x1)**2 + (y - y1)**2 < r1**2 >>> mask_circle2 = (x - x2)**2 + (y - y2)**2 < r2**2 >>> image = np.logical_or(mask_circle1, mask_circle2)
接下來,我們想要分隔這兩個圓形。我們在與背景距離的最大值處產生標記
>>> from scipy import ndimage as ndi >>> distance = ndi.distance_transform_edt(image) >>> from skimage.feature import peak_local_max >>> max_coords = peak_local_max(distance, labels=image, ... footprint=np.ones((3, 3))) >>> local_maxima = np.zeros_like(image, dtype=bool) >>> local_maxima[tuple(max_coords.T)] = True >>> markers = ndi.label(local_maxima)[0]
最後,我們在影像和標記上執行分水嶺
>>> labels = watershed(-distance, markers, mask=image)
此演算法也適用於 3D 影像,例如,可用於分隔重疊的球體。